一年前に読んだ論文に再挑戦してみる
更新履歴
2019/06/17: GPUによる学習結果(conll)を追記、他修正
2019/06/18: GPUによる学習結果(CHEMDNER)途中経過を追記
2019/06/20: GPUによる学習結果(CHEMDNER)途中経過を追記
2017年6月頃にHoloLensアプリの開発を始めました。ちょうどこのタイミングでTECH LAB PAAKに10期として応募したみたいです。
2017年10月ごろにVCと相談しながら自然言語処理で論文からの情報抽出に挑戦しようとなったようです(メッセンジャーの履歴を辿ると)。
そこから自然言語処理を一から勉強し始めて、(入門自然言語処理とか)
友人伝いにNLPをやっている人にアドバイスを聞きながら、自分がやろうとしているタスクがNamed entity recognitionという名前のタスクであることを発見しました。
ある意味ではここまでが一番大変でした。なんてググっていいかすらわからなかったので。
別の意味ではここからはもっと大変でした。
とりあえず、NERのベンチマークにはCoNLL 2003が使われていること、Lample 2016のBi-directional LSTM CRFのモデルが精度高いことなどにたどり着き、
Bi-directional LSTM CRFを理解するために、Bi-directional LSTMを理解するために、LSTMを理解するために、RNNを理解するという一歩一歩必要な知識を後からつけていった目的志向の歪な知識体系が組み上がりました。
その後学科の領域プロジェクトという実習?で松尾研に配属。2018年6月のこと。すでにこの時点で一年近く経っていました。
配属期間中にLampleのBi-LSTM CRFによるNERを実装しようと計画しますが全然無理で、コードを読み解いてCHEMDNERのデータで学習させるだけで精一杯でした(論文のabstractから薬剤名を抽出しようとした)。
その時の結果がこれ。
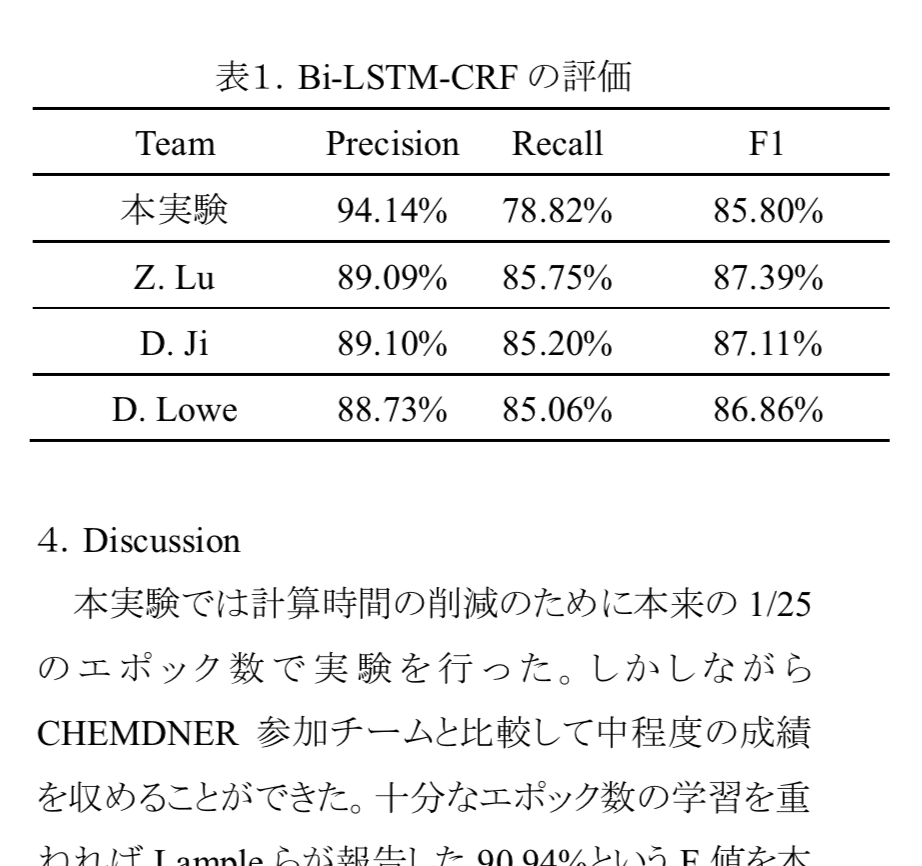
薬剤名を深層学習で NERできそうという確信は持てたものの、全く実装力が足りず、コードも理解できていない無力さ。
白状すると全然コード追えてませんでした。論文で概要は理解したようになって、著者による親切なGithubに頼ってなんだか実験の真似事をやってみたって感じでした。
ちなみに上のレポートでトータルの1/25(= 4 epoch)で止めたって書いてますが、GPUインスタンスのストレージがログファイルで逼迫されて学習が途中で止まったというのが正確な表現です。見返すと言い訳がましくて恥ずかしい。
時間がなくてやり直しができなかったので、計算時間削減したってのは事実ですが。
で、今回のお題は理解があやふやだったLampleのコードを今一度ちゃんと理解しようという試みです。
ちょうど一年ぐらい経つわけですが、どこまで読めるようになってるか。
目次
論文内容のおさらい
以前まとめました。が、読み込みが足りないので後ほどより綺麗にまとめます。
TODO: まとめ直し
リポジトリ概観
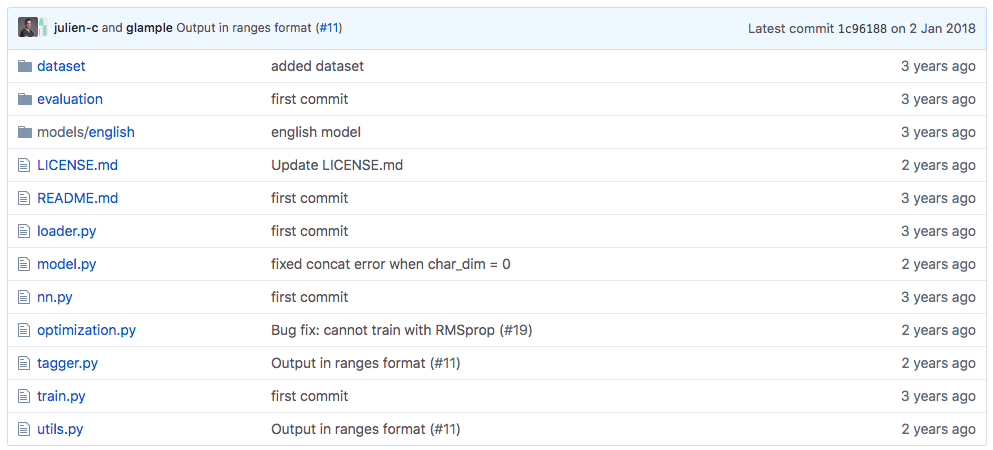
だいたいこんな感じです。
- dataset/: データセット
- evaluation/: 評価用関数
- models/: 学習済みモデルのパラメータ
- loader.py: データを読み込む
- model.py: モデル全体の構成
- nn.py: 各層の定義
- optimization.py: 最適化
- tagger.py: 学習済みモデルを使ってタグ付け
- train.py: 教師データを使って学習
- utils.py: その他
メインはtagger, train, model, nnを読めば大体掴めそうです。
環境構築
Githubのページをfork、自分のリポジトリで管理します。
まずはローカルに持ってきて挙動をチェックします。
git clone https://github.com/glample/tagger.git
Python 2.7(!)対応なのでpipenvで仮想環境を管理します。
pipenvでPython 2.7環境を用意、起動。
requirements.txtもないので、必要な外部パッケージは逐一入れていかなくてはいけません。
pipenv install --python 2.7
pipenv shell
pipenv install theano numpy
推論: tagger.py
初めから英語の文章に対しては学習済みモデルがリポジトリに入っています。これを使ってタグ付けしてみましょう。
推論時に指定できるパラメータは5つ
- model: 学習埋みモデルのディレクトリ
- input: 入力ファイル
- output: 出力ファイル
- delimiter: 単語とNERタグを繋ぐデリミター
- outputFormat: 出力ファイル形式(jsonかその他)
入力ファイルは一行に一文、トークナイズしておいてとのこと。
The input file should contain one sentence by line, and they have to be tokenized.
https://github.com/glample/tagger
適当なファイルをinput.txtとしましょう。
ディレクトリの見えるところにファイルたくさん生成したくないのでdataset/に突っ込んでおきます。
echo 'Harry Frederick Harlow was an American psychologist best known for his maternal-separation, dependency needs, and social isolation experiments on rhesus monkeys, which manifested the importance of caregiving and companionship to social and cognitive development.' > dataset/input.txt
Harry Frederick Harlowが人名と認識されるといいですね。
実行。
python tagger.py --model models/english/ --input dataset/input.txt --output dataset/output.txt
Loading model...
Compiling...
Tagging...
---- 1 lines tagged in 0.2237s ----
完了(多少警告文が出たけど省きます)。
出力ファイルを見てみます。
Harry__B-PER Frederick__I-PER Harlow__I-PER was__O an__O American__B-MISC psychologist__O best__O known__O for__O his__O maternal-separation,__O dependency__O needs,__O and__O social__O isolation__O experiments__O on__O rhesus__O monkeys,__O which__O manifested__O the__O importance__O of__O caregiving__O and__O companionship__O to__O social__O and__O cognitive__O development.__O
Harry__B-PER Frederick__I-PER Harlow__I-PERが人名として認識されていました。AmericanもMISC(その他)の固有表現と勘違いされていますが、使い方は良さそう。
ちゃんと動く。
学習: train.py
では本番、学習を回してみます。
オプションはこんな感じ。all_embとcap_dimだけよくわからなかったのでコードを追いましたが、だいたいわかりやすい。
- train, dev, test: それぞれのデータ
- tag_scheme: タグのフォーマット(IOB or IOBES)
- lower: 小文字化
- zeros: 数字を0に変換
- char_dim: 文字ベクトルの次元
- char_lstm_dim: 文字LSTM層の次元
- char_bidirect: 文字LSTM層を両方向にするか
- word_dim: 単語ベクトルの次元
- word_lstm_dim: 単語LSTM層の次元
- word_bidirect: 単語LSTM層を両方向にするか
- pre_emb: 事前学習のembedding
- all_emb: ???
- cap_dim: ???
- crf: CRF層を使うか
- dropout: ドロップアウト率
- lr_method: 学習率とメソッド
- reload: 最後に保存したモデルを読み込むか
では始めからdataset/ に入っていたeng.train, eng.testa, eng.testbを使って学習を回してみます。
ちなみに学習に使うデータはこんな形式。
列はtoken, pos tag, chunk tag?, ner tagのようです(cunkingやったことないので3列目は怪しい)。
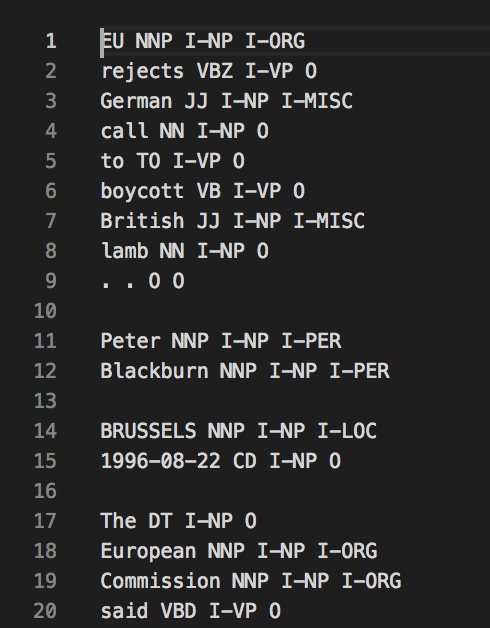
とりあえず動くか確認したいので、char_dim=3, char_lstm_dim=3, word_dim=3, word_lstm_dim=3で指定。
train.pyのn_epochsも10に変えて時短を図ります。
python train.py --train dataset/eng.train --dev dataset/eng.testa --test dataset/eng.testb --char_dim 3 --char_lstm_dim 3 --word_dim 3 --word_lstm_dim 3
Model location: ./models/tag_scheme=iobes,lower=False,zeros=False,char_dim=3,char_lstm_dim=3,char_bidirect=True,word_dim=3,word_lstm_dim=3,word_bidirect=True,pre_emb=,all_emb=False,cap_dim=0,crf=True,dropout=0.5,lr_method=sgd-lr_.005
Found 23624 unique words (203621 in total)
Found 84 unique characters
Found 17 unique named entity tags
14041 / 3250 / 3453 sentences in train / dev / test.
Saving the mappings to disk...
Compiling...
Starting epoch 0...
/Users/Ryota/.local/share/virtualenvs/tagger-lample-DsxM51Rj/lib/python2.7/site-packages/theano/tensor/subtensor.py:2197: FutureWarning: Using a non-tuple sequence for multidimensional indexing is deprecated; use `arr[tuple(seq)]` instead of `arr[seq]`. In the future this will be interpreted as an array index, `arr[np.array(seq)]`, which will result either in an error or a different result.
rval = inputs[0].__getitem__(inputs[1:])
50, cost average: 17.655391
100, cost average: 13.072090
150, cost average: 13.408389
200, cost average: 13.048931
250, cost average: 11.731661
...
とこんな感じで学習が進みます。
学習過程のスコアは以下のような形で見ることができます。
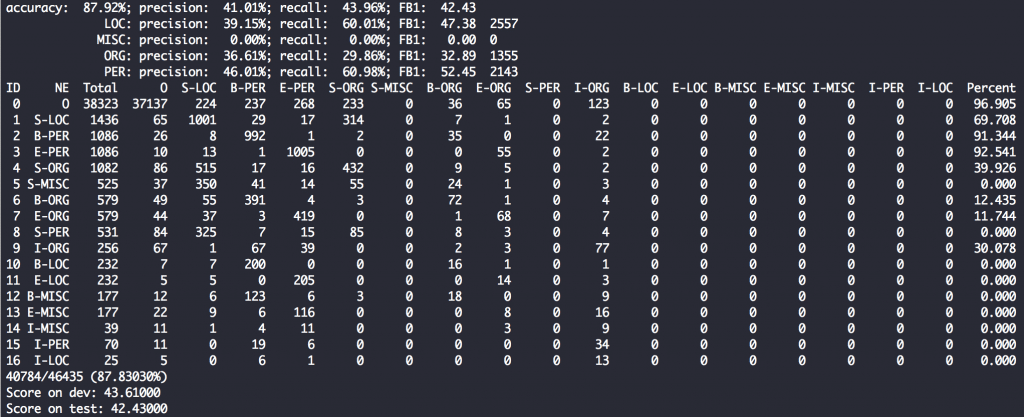
上の5行は全体および各エンティティタイプに対するprecision, recall, f1 scoreを出しています。
下のマトリックスは各エンティティタグがどのように予測されたかが一目でわかるので、どのように間違えがえがちかの傾向が掴めます(対角線状が正解)。
例えば上の画像ではS-MISC(単体のその他のエンティティ)は525ありますが、そのうち350がS-LOC(地名)と誤解されていることおがわかります。
一方でB-PER(人名の先頭)は1086中992が正解していて正解率91.3%と高い精度で予測ができているようです。
1時間弱で完了。超適当な学習でしたが、参考までにスコアを出します。
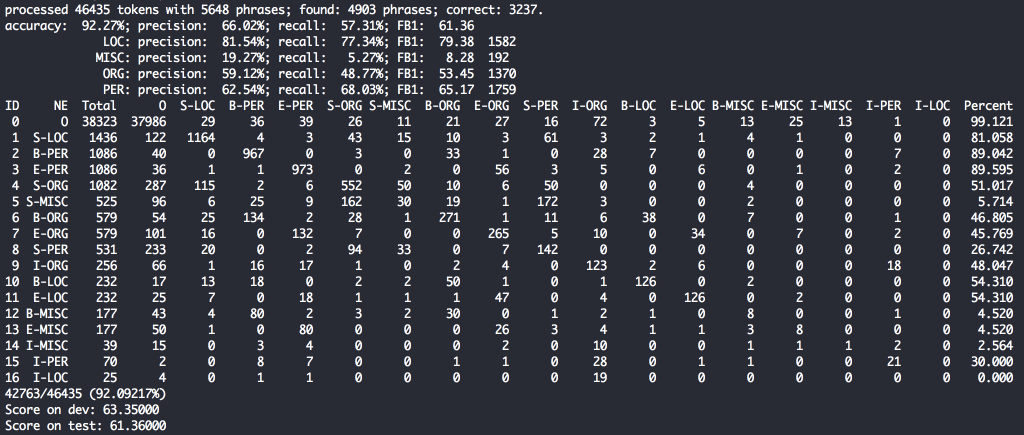
testで61.3%。
GPUで学習
ではGPUで本気モードやってもらいます。
ところで、CPUで動かした時はpipenvで環境構築したのですが、GPUを使おうと思ったら詰まったので(後述)、ここからはPython環境はanaconda、パッケージマネージャはcondaを使います。
conda createでpython 2.7環境を構築。
conda create -n py27 python=2.7
source activate py27
conda install -c mila-udem -c mila-udem/label/pre theano pygpu
公式でRECOMMENDEDされている方のインストール方法をとりました。
ちなみに通常のconda installでtheanoを入れると1.0.3が入るのでGPU利用でエラーが出る(後述)。
エラーに苦しむ図
通常のconda install theanoをするとimport theanoだけでエラー出る。
AttributeError: ('The following error happened while compiling the node', DnnVersion(), 'n', "'module' object has no attribute '_get_ndarray_c_version'")
調べるとこちらのissueがヒット。
theano 1.0.4にアップデートしろとのこと。お気をつけあそばせ。
気を取り直して。theanoでGPU使う時はこんな感じ。環境変数で指定します。
THEANO_FLAGS=mode=FAST_RUN,device=cuda,floatX=float32 python yourscript.py
Lampleのコードを動かすには、こんな感じ。なっがい!
THEANO_FLAGS=mode=FAST_RUN,device=cuda,floatX=float32 python train.py --train dataset/eng.train --dev dataset/eng.testa --test dataset/eng.testb
さらにtimeコマンドをつけて学習にかかった時間を見ます。さらにさらにnohupコマンドをつけることで、ssh接続切ってもサーバで勝手に計算を続けてくれます。最後にリダイレクトでout.logに標準出力を出します。
naga-i !
THEANO_FLAGS=mode=FAST_RUN,device=cuda,floatX=float32 time nohup python train.py --train dataset/eng.train --dev dataset/eng.testa --test dataset/eng.testb > out.log &
(標準出力は処理が終わるとまとめて出力されるので、途中経過をtailコマンドで見られないのどうにかしたい)
ちなみにホームディレクトリに.theanorcという設定ファイルを置くことで長い環境変数の部分は省略できます。
vi ~/.theanorc
[global]
floatX = float32
device = cuda
設定ファイルの細かい項目は公式を。
プロセスが動いているかどうかはpsコマンドで確認できます(timeと別個扱いになる?)。
ps aux | grep train

(1行目の950分のプロセスは先に起動した方。実は時系列的に次のnvidia-smiでGPUをチェックしたのが始まりで、GPU使ってなくない?となってこれまでのGPU利用する方法の模索に戻った次第)
GPUを使用しているかどうかはNVIDIAのドライバを使っている場合、nvidia-smiコマンドで確認できます。何秒か置きに確認したければ、-lオプションを使います。
nvidia-smi -l 5
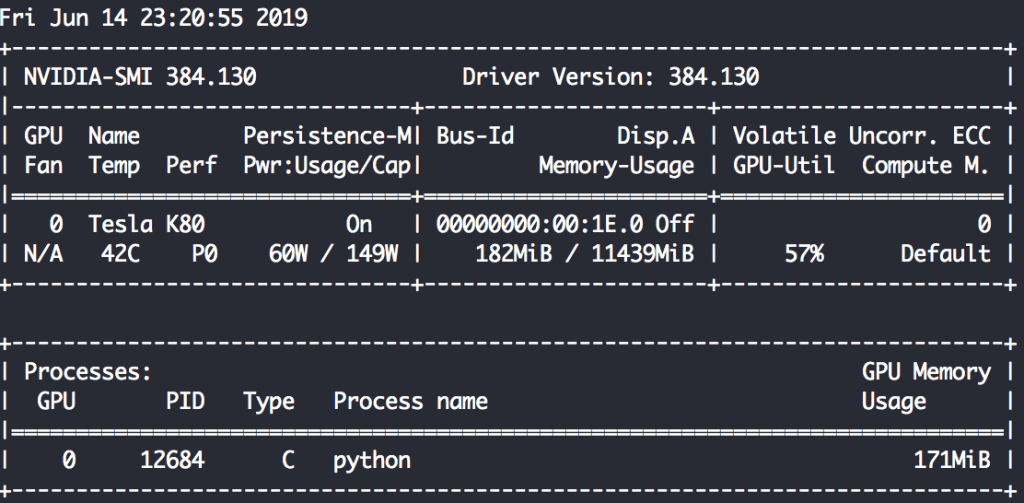
使ってますね!…使ってますね!!
一般にRNNはGPUの恩恵をあまり受けないと言いますが、どうなんでしょう。
以前回した時は本コードが吐き出す中間生成ファイルにストレージが逼迫されて途中で学習が止まってしまったんですが。。。
今回は残り23GBあります。安心して寝れるでしょう。
結果が出たら更新します!
(一晩経って)
ログを見たところ、750 min (12.5 h)で21 epochでした。
今回はデフォルト設定で回したので全100 epoch。あと丸2日くらいかかりますね。
ちなみに現段階でtestデータに対してベストな結果を出したモデルはこんな感じのスコア。
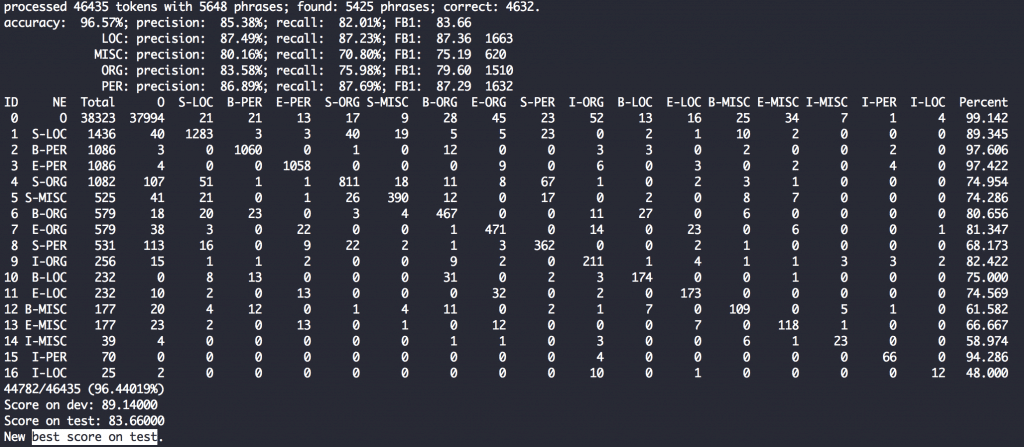
以前出したレポートで4epochでこんな精度出ると書いたけど、本当だったかドキドキしてきました。
エンティティが化学物質しかないから精度が高かった、と信じたい。間違ってたら謝ろう。

一方、CPUでも回していますが、こちらは3200 min (53 h)。ただ吐き出してるログファイルが壊れて読めなくなっていたため、残り時間不明。。。
学習終了
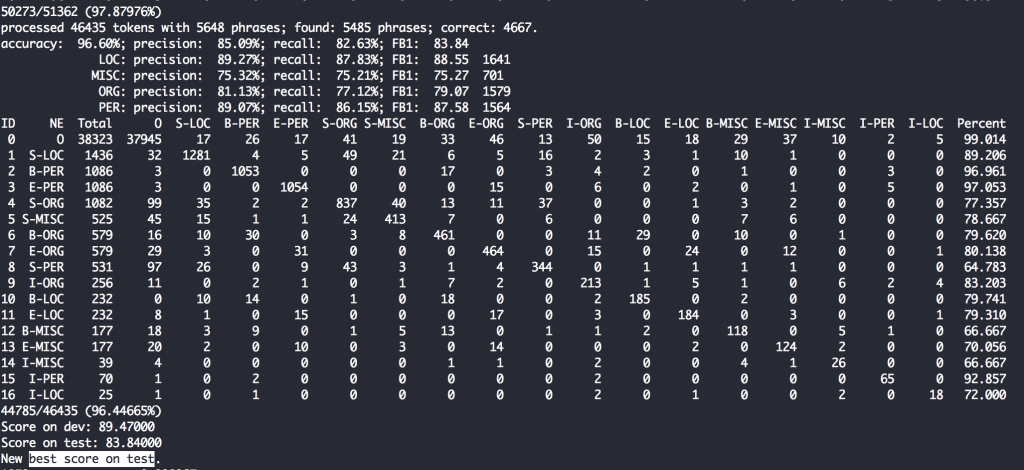
一番testの結果がよかったモデルでスコアは83.84%。
論文内で標榜していた90.94には遠く届いていません。全部デフォルト設定でやったからそこら辺のパラメータを最適化しないといけないんだろうか。デフォルト設定が最適にしてくれれば良いのに。
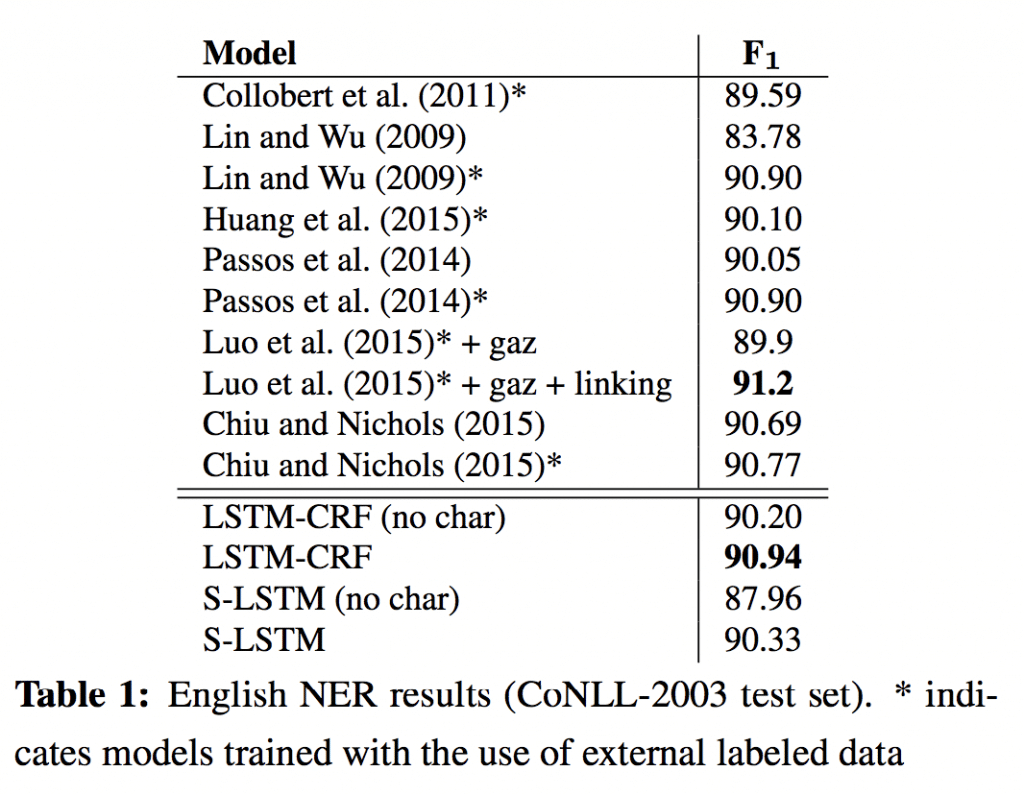
ひとまず、結果には疑問が残りますがGPUで3日ほど回して学習が終了しました。CPUで学習させているプロセスは196時間=8.2日経っても終わっていないので、GPU使った恩恵はありましたね。
CHEMDNER corpusで学習
CHEMDNERについて
CHEMDNERは2012年にBioCreative IVで行われたshared taskです。
2つのタスクが設定され、chemical entity mention recognition (CEM)では論文中の化学物質の名称を検知することを目的とされました。
コーパスはこちらから公開されています。大まかな情報は論文でまとめられています。
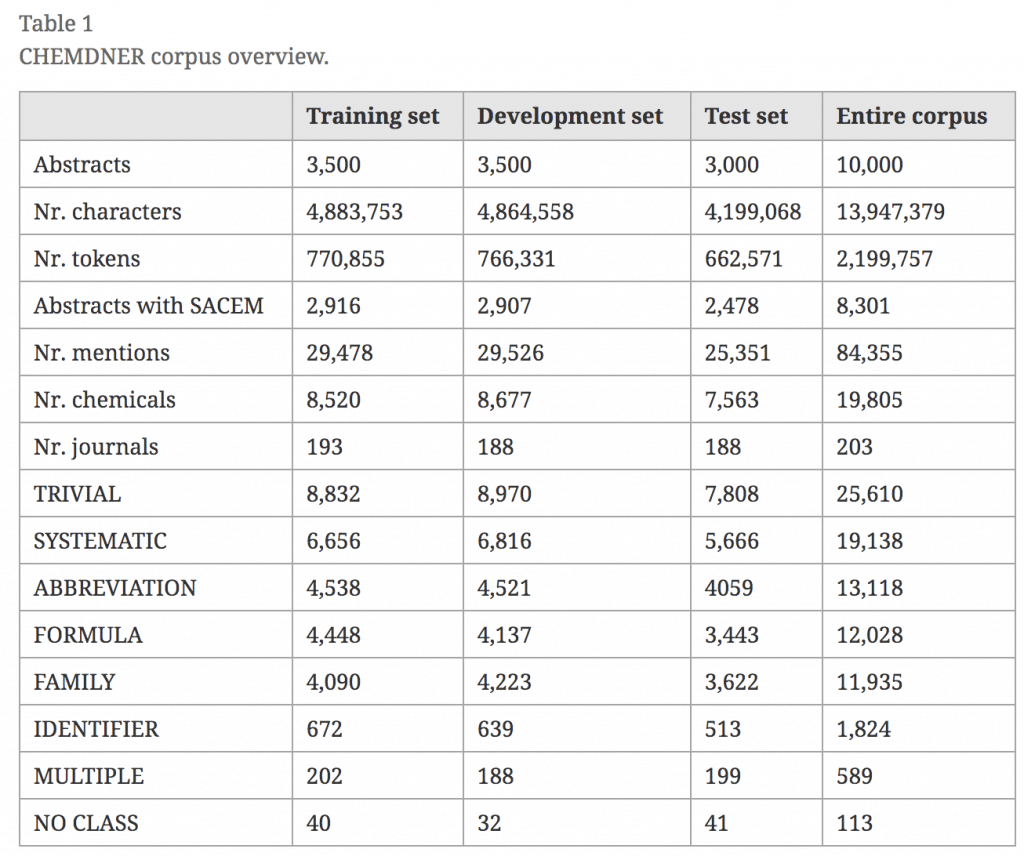
また結果も別の論文でまとめられています。この表を見ると2012年時点ではF1スコア87.4%で優勝できることがわかります。
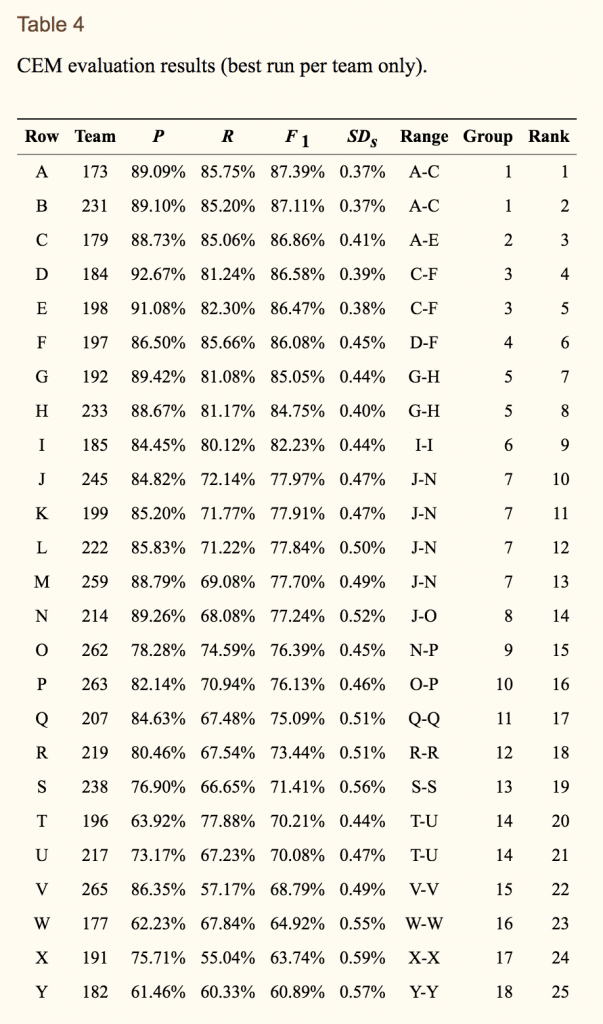
データ形式
さて実際にコーパスを開いて見てみます。
evaluation.abstracts.txtにはpaper id, title, abstractがタブ区切りで保存されています。

そしてevaluation.annotations.txtには各論文に含まれる化学物質名のアノテーション情報が記載されています。
列は左から、paper id, title/abstract, strat, end, name, typeです。こちらもタブ区切り。
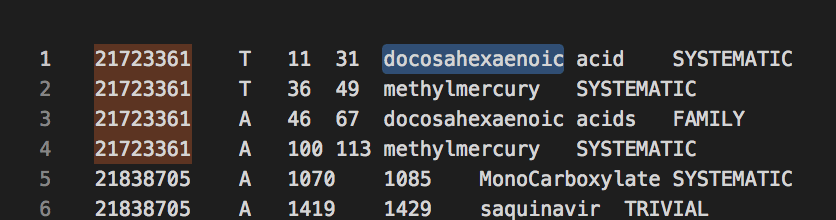
このような形でテキストとアノテーションが別ファイルにまとめられていてpaper idで紐づける必要があります。
このテキストファイルとアノテーションファイルがtrain, dev, testにそれぞれtraining.xxx.txt, development.xxx.txt, evaluation.xxx.txtというファイル名で用意されています(よって今回使うのは合計6ファイルです)。
おさらいですが、今回のLampleのコードに突っ込むにはこんな形でなくてはいけません。
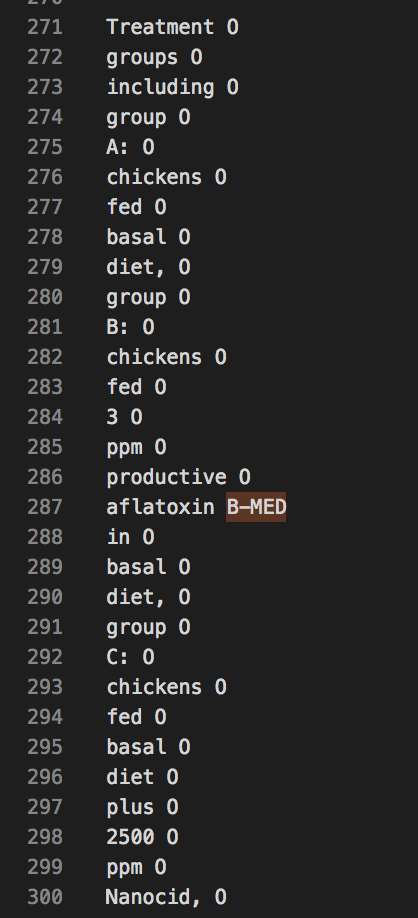
したがって、ちょろっとしたコードを書いてxxx.abstracts.txt, xxx.annotations.txtを上記形式のファイルを生成します。
こうして出来上がったファイルをtraining.chem.txt, development.chem.txt, evaluation.chem.txtと称して、Lampleのコードのインプットにします。
前項「GPUで学習」で開始した学習がまだ終わっておらず、GPUの使用率が50%ちょっとであったことから、CHEMDNERモデルも同時に回せるのでは?と思いました。
しかし学習開始しても使用率が変わらず。100%使い切ってくれることを期待したんですが。
周りの人に聞いたところ、以下の記事のようにマルチプロセスにしなくてはいけないらしい。また新たなことを学んだ。次回から活かしたい。
学習開始
前項の学習が3日ほど経って終わったので回すことにします。
環境変数は~/.theanorcに埋め込み、さらに学習過程をComet.mlに飛ばすことにしたのでこっちでログを記録する必要も無くなりました。
nohup python train.py --train dataset/training.chem.txt --dev dataset/development.chem.txt --test dataset/evaluation.chem.txt &
sukkiri !
(Comet.mlの使い方に関しては別途まとめます。)
ではまた学習結果が出た頃に更新します。
1 epoch経って
文量に差があったためか1 epochにだいぶ時間がかかりました。
testで79.02%。やはりエンティティが1つな分精度が高いですね。
一晩経って
4epochでtestスコア84.44%。すでにCoNLLデータの100epoch時testスコアを超えました。
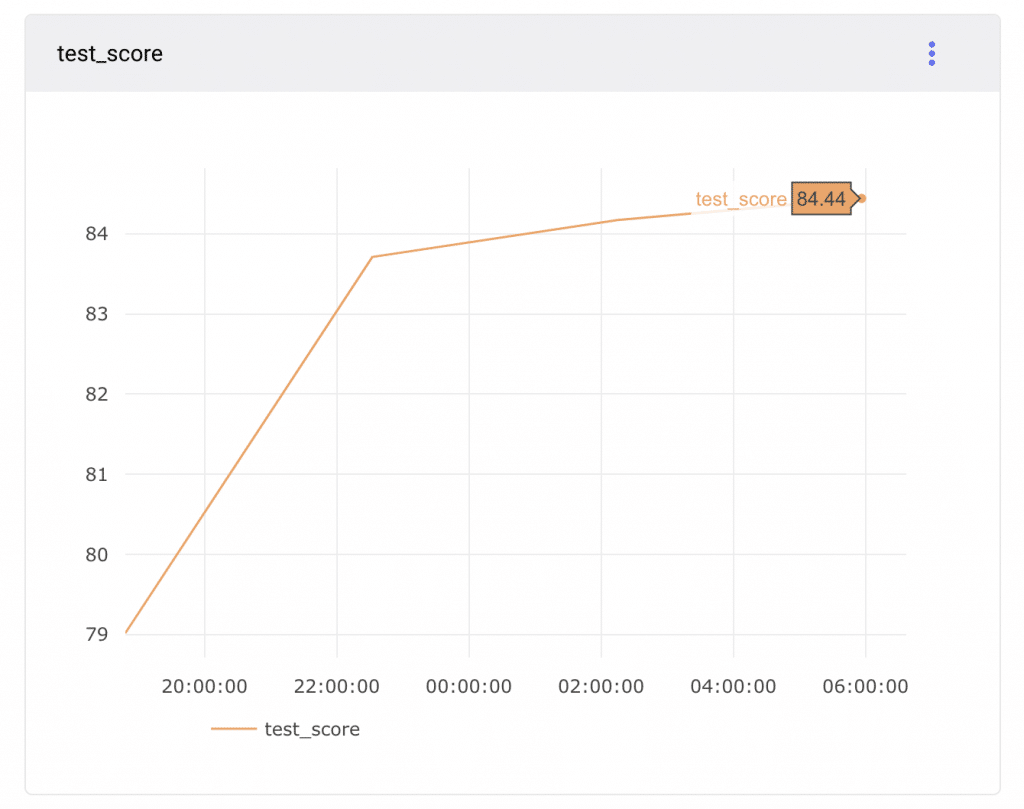
前回大学のレポートで報告した結果が同epoch数で85.80%でした。んー、まぁありえなくなくはないのか?とりあえず大きな過失はなかったと言っても良さそう(大丈夫?)でよかった。自分が間違った学習方法で出た結果をレポートにしていなさそうで良かった。
学習終了
TODO: 前項の学習が終了したら学習開始
1日で4 epochなので25日(7月中旬になる予定)。遠い!
比較
文量とエンティティ数に対して学習時間と精度がどれくらい変わるのか知りたいですね。参考というほどにもなりませんが、簡単に比較しようと思います。
| データセット | CoNLL 2003 | CHEMDNER |
| 文量(train+dev+test) | 22,134 | 71,498 |
| エンティティ数 | 4 | 1 |
| 学習時間 | 約3日 | 推定25日 |
| スコア(test) | 83.84% | ??? |