macOSでBratを使う
以前生命科学論文のアノテーション済み公開データを使って薬剤のNamed Entity Recognitionをやりました。
Relation Extractionを試みるにあたって自分でアノテーションをやることにしました。
理想としてはこんな感じです。
Bratとは
ウェブベースのアノテーションツールです。ブラウザ上で簡単に操作できます。
特に構造的なアノテーションに特化しているとのことです。
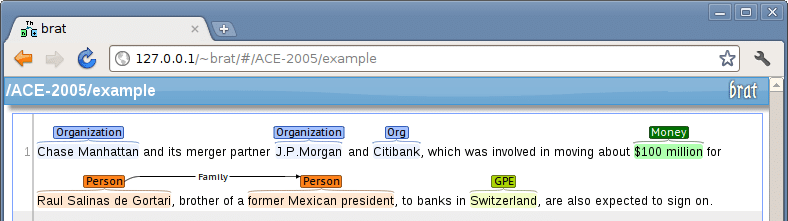
基本的に2つのアノテーションができます。
- text span annotations: 「$100 million」が一つのまとまりに対して「Money」というタグ付け
- relation annotations: 「Raul …」と「former …」の間に「Family」という関係性をタグ付け
サイトでオンラインデモができます。
インストール
以下の環境にBratをインストールしました。
- MacBook Pro (15-inch, 2018)
- macOS High Sierra 10.13.6
- Python 2.7.11
Python2系が推奨されているので注意します。またJSONサポートの関係で2.6以上をお勧めします。
トップページからファイルをダウンロードします。
展開します。
tar xzf brat-VERSION.tar.gz
ディレクトリ内に移動。
cd brat-VERSION
作業用ディレクトリを作成。
mkdir -p data work
コンフィグファイルをコピーします。
cp config_template.py config.py
ユーザとパスワードを追加しておきます。
vi config.py
USER_PASSWORDの部分に辞書形式で追記。

とりあえず簡易サーバで確認します。
python standalone.py
ウェブブラウザからhttp://127.0.0.1:8001にアクセスします。
以上です。
インストールについてはこちらを参考にしました。
ここではMacのローカルで動かしましたが、Liunxならinstall.shを実行するだけで簡単にセットアップができるとんことです。
Windowsなら仮想マシン立てるのが推奨されていました。
簡易サーバはセキュリティ不十分なので本番で動かす時はApacheとか使ってねーとのことでした。気をつけましょう。
動作を確認する
先ほどのTutorialをOKするとファイルの選択画面が表示されます。
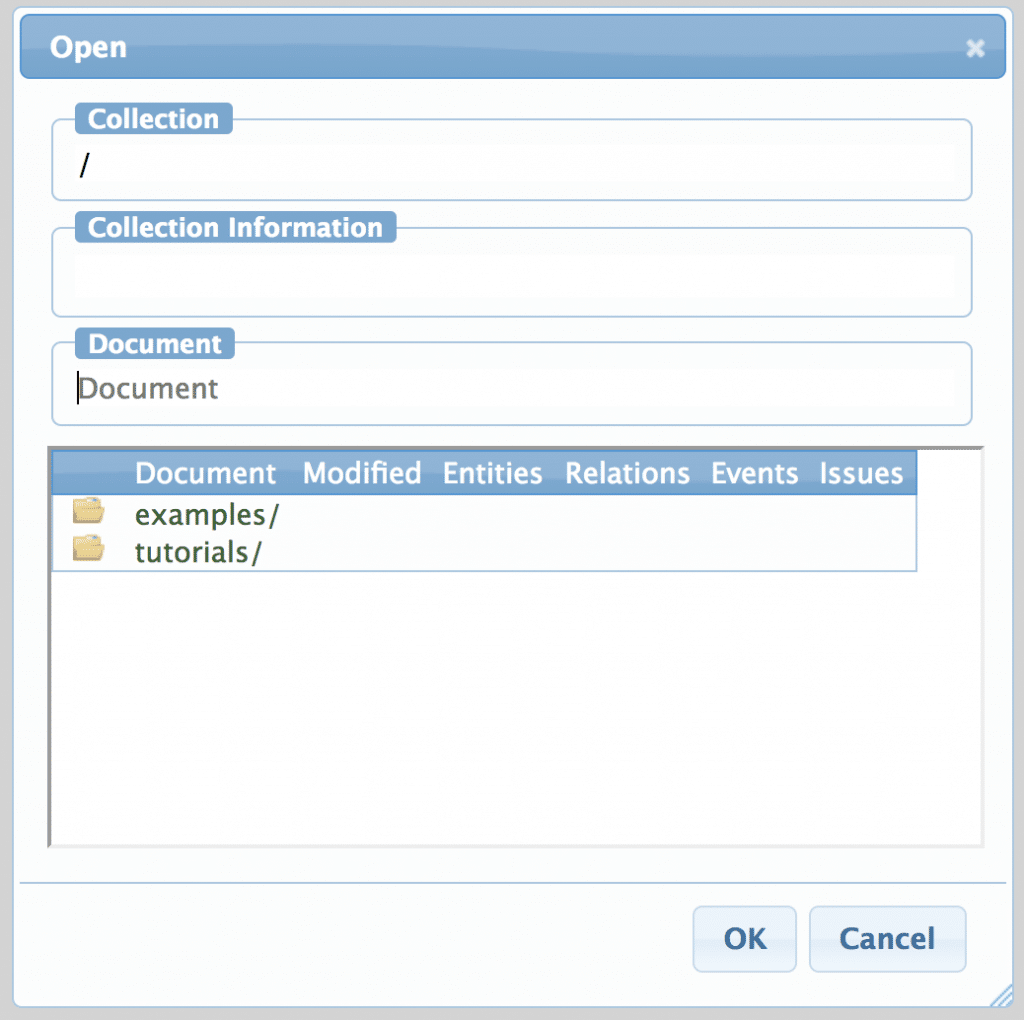
tutorials/ を選択するとさらにそのディレクトリ内が表示されます。
どんどんクリックしてtutorials/tutorials/bio/000-introductionを開きます。
こんな感じでアノテーション済みのファイルが開かれます。
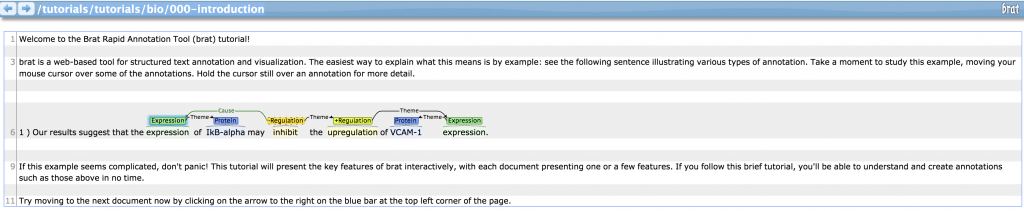
アノテーションを追加するにはログインする必要があります。
上部のバーにカーソルを合わせるとメニューが開くので右のLoginをクリック、先ほどconfig.pyに設定したユーザ名とパスワードを入力します。
ログイン状態でテキストをドラッグするとアノテーションウィンドウが開きます。
まずはtext span annotationを試します。
適当に「structured text」の部分をドラッグして、Proteinを割り当てます。
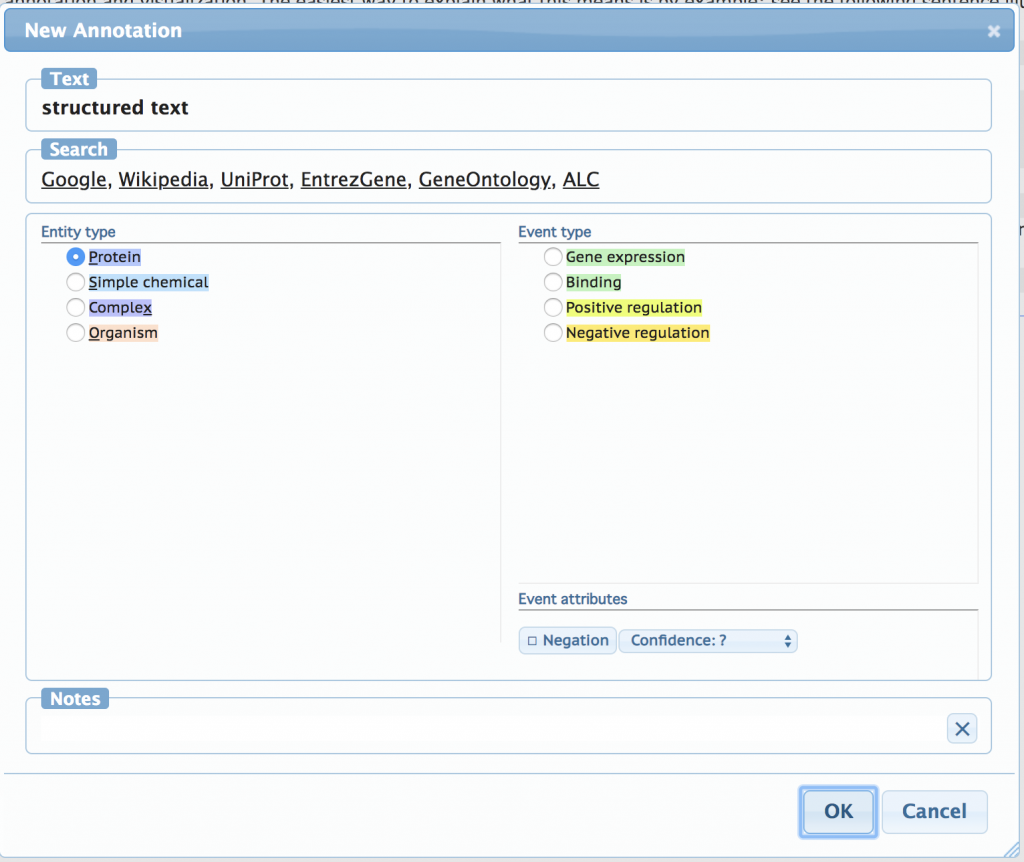
ついでに「visualization」にGene expressionをタグ付けします。
こんな感じでアノテーションが追加されました。

次にrelation annotationを行います。
「visualization」から「structured text」にドラッグ。
relation用のアノテーションウィンドウが開くのでThemeを選択します。
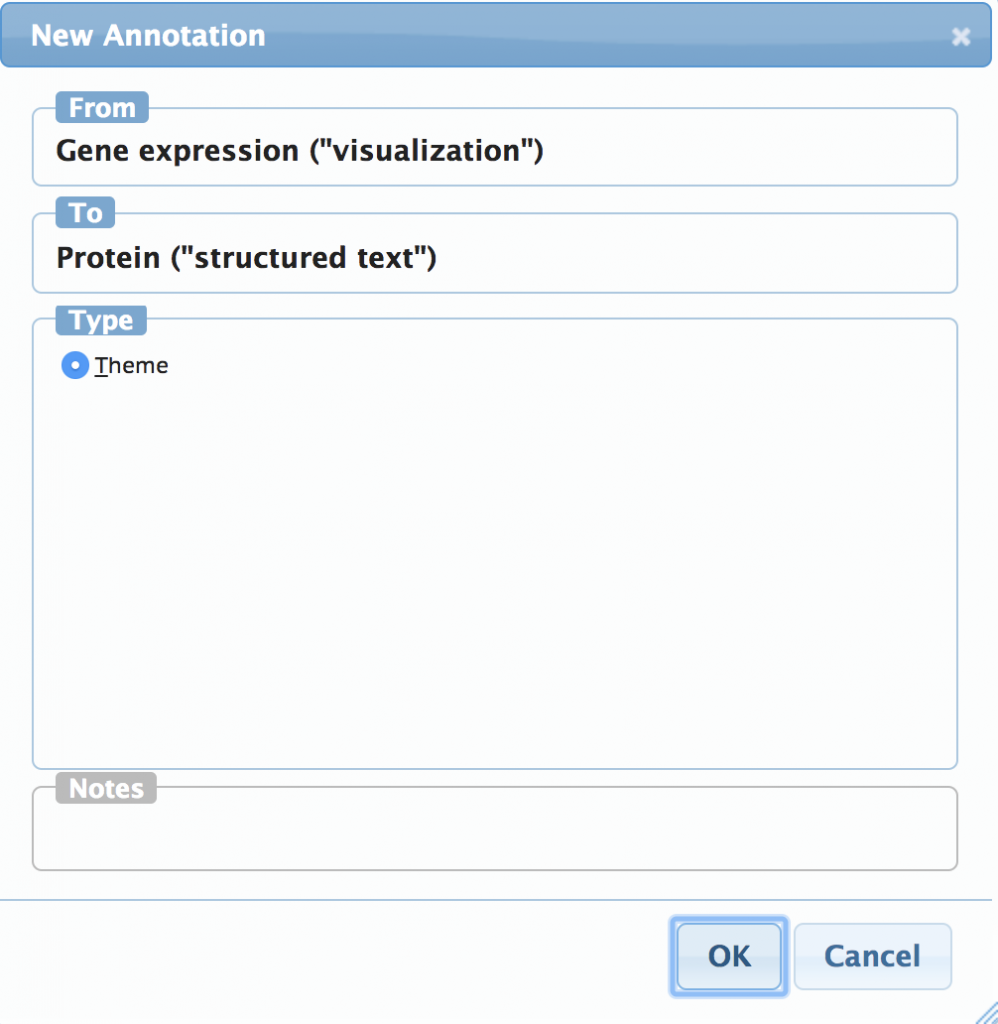
こんな感じ。

アノテーションを変更・削除したい時はタグをダブルクリックするとウィンドウが開きます。
以下公式を参考にしました。
おまけ
大まかに使い方がわかったので、頑張ってアノテーションします。
設定ファイルについては以下の記事でまとめました。