EXACT2:生命科学の実験プロトコル用オントロジー
生命科学実験の自動化や再現性担保のためにプロトコルを機械可読にすることは大いに意義があります。
しかし現在多くの実験プロトコルは自然言語による自由記述でコンピュータにとっては扱いづらい形式です。
EXACT2は実験プロトコルをセマンティックに表現するためのオントロジーです。
ここでは主にEXACT2の論文の内容を紹介し、その特徴を概説します。
目次
EXACT2: the semantics of biomedical protocols
書誌情報は以下の通りです。
- Title:EXACT2: the semantics of biomedical protocols
- Author:Larisa N SoldatovaEmail author, Daniel Nadis, Ross D King, Piyali S Basu, Emma Haddi, Véronique Baumlé, Nigel J Saunders, Wolfgang Marwan and Brian B Rudkin
- Journal:BMC Bioinformatics, 2014
- Date:2014/12/27
なお、EXACT2は著者らが以前に発表したEXACTをアップデートしたオントロジーです。
概要
生命科学の実験プロトコルを以下の3つの要素で表します。
- action:「heat, incubate, …」などの動作
- descriptor:「entity, temperature, period, …」などのactionの条件を表現する記述子
- relation:「is-quality-of」などactionとdescriptorの関係
記述法としての特徴:OBI, Tavernaとの比較
生命科学のプロトコルを機械可読に記述する需要は大きく、いくつもの先行研究があります。
Ontology for Biomedical Investigations(OBI)
OBIは生命科学の現象を記述することを目的としています。
しかし実験プロトコルという観点からは必要な情報が網羅されていなかったり(例えば試薬を何℃で保存するか)、逆に表現が複雑すぎて(entityにいくつものタイプがタグ付けされる)組合せ爆発を起こしたりする課題がありました。
EXACT2では思い切ってタグの数を減らし、現実的に機械で解析することにフォーカスしました。
どちらが優れているかではなく、以下のように目的が違います。
- OBI: 生命科学の現象にオントロジーを導入したい
- EXACT2: 実験プロトコルを簡潔に過不足なく表現したい
そのためEXACT2ではentityのタイプがbiochemical entityやequipmentぐらいざっくりしています。
(OBIではblood specimenやparaffin storageまで細かく分類する)
Apache Taverna
Tavernaは特定ドメインのワークフローを記述することを目的としています。
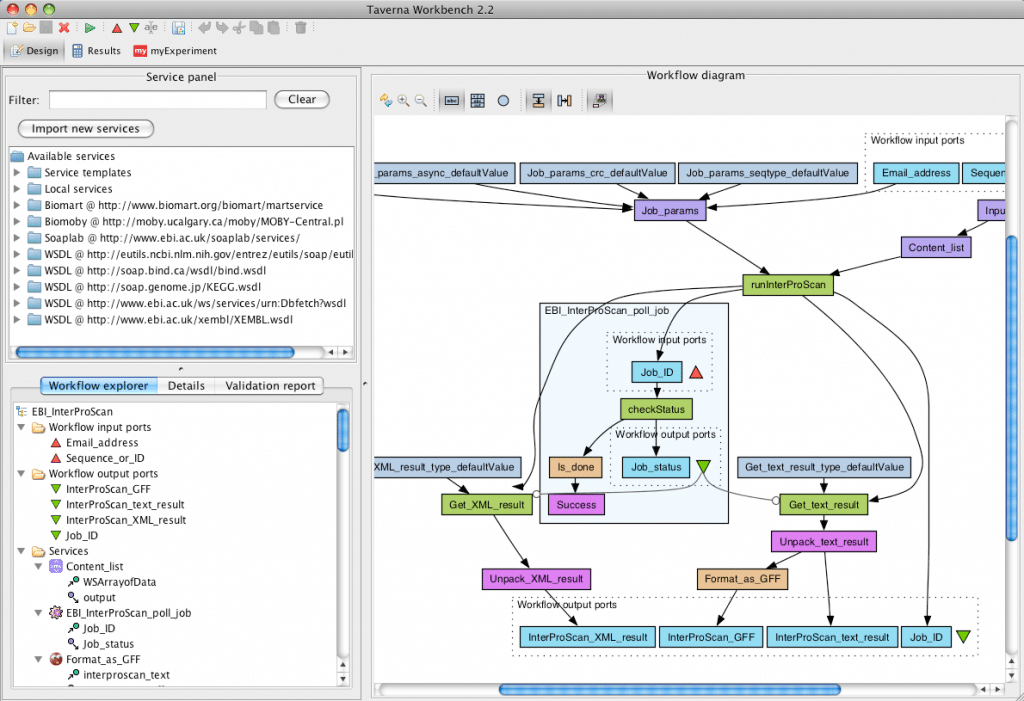
以下の図のようにentity同士を繋いでワークフローを構築します。
出展: Apache Taverna
実験プロトコルを入力する場合は「何」を行なったかに着目してワークフローに落とし込みます。
しかし「どのように」行われたかという情報を十分に記述できないそうです。
EXACT2はhowの部分を補完する記述形式であり、より生命科学のドメインに適しています。
(例えばstoreというactionに対してtemperatureやperiodなどのdecriptorがhowに当たります)
EXACTからの追加内容
プロトコル中には必ずしも全ての情報が記述されているわけではなく、読み手の解釈に任せられる部分があります。
例えば0.1% BSA(ウシ血清アルブミン)と書かれている場合、その溶媒は純水やPBSなどが考えられます。
他にも滅菌状態などの条件は(文脈によっては)暗黙の了解として省略されることがあります。
しかしこのような状態はコンピュータに読ませるには不適切です。
そこでEXACT2では生命科学の専門家がどのようなdescriptor(記述子)が必要かあるいはあってもなくても良いかを判定しました。
(descriptorにはtemperature, equipmentなど)
例えば「storeというactionに対してはtemperatureの情報が必要ですが、filterというactionには必ずしも必要ない」などです。
複数のオントロジーを取り入れて構成
EXACT2はMIREOT(Minimal Information to Reference an External Ontology Term)という原則に従い複数の外部オントロジーの必要な部分をインポートして構成されます。
entityにURIを振る際、生命科学のオントロジーや実験器具のオントロジーを参照しているということです。
複数のオントロジーをインポートするのにはOntoFoxというウェブアプリケーションを利用しているとのことです(初めて知ったのであとで調べます)。
実例
前置きが長くなりましが、実際にEXACT2を使ってプロトコルを半自動的にアノテーションする流れを紹介します。
- テキスト形式のプロトコル、EXACT2と手がかり(「℃」はtemperature、「in order to」はgoalに関する、など)を入力
- 研究室特有の情報を入力(実験器具、薬剤、略語など)
- テキストを処理
- named entityを同定、標準化
- 名詞、動詞などを同定
- 文を区切る(Genia Sentence Splitterを利用)
- experimental actionを同定
- 動詞からactionを同定
- 一つの文に複数のactionが見つかったら文をコピーして別々に記載(一つの文にactionは一つになるように)
- descriptorを同定
- 入力した手がかりからactionの条件であるdescriptorを同定
- 必須のdescriptorが欠けていたら人手で入力
- action, descriptorとその値を出力
- ユーザが出力をチェック、修正
- 抜けがあれば追加
以上の流れを図で表すと以下のようになります。
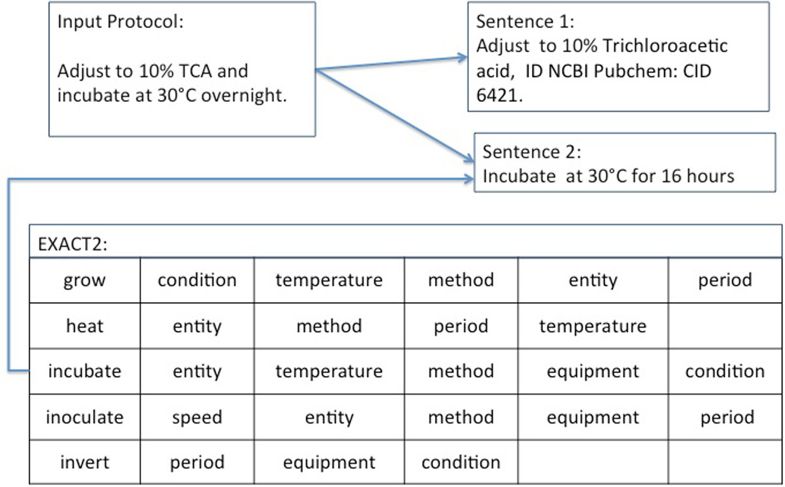
まず自然言語からactionを見つけます(grow, heat, …)。
論文執筆時点で83-95%のactionを見つけられるそうです。
右側に必要なdescriptorが表記されています(growに対してはcondition, temperature, …)。
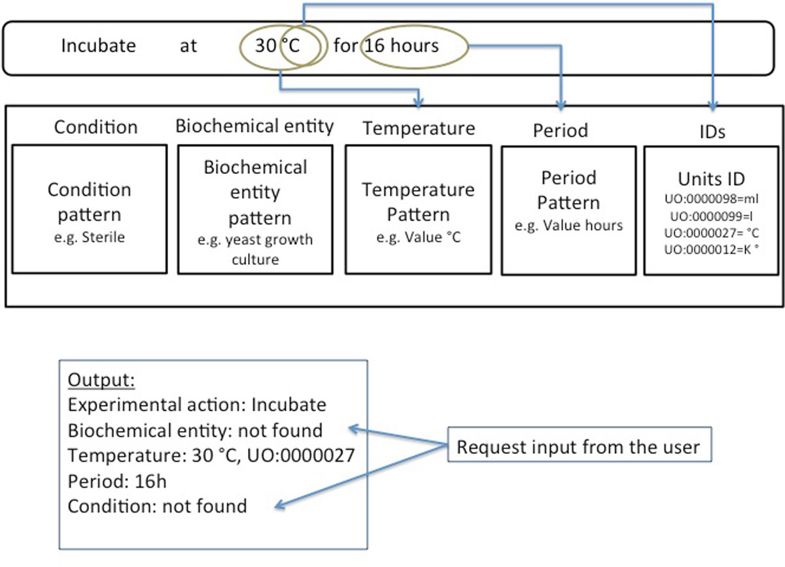
次にdescriptorを文章から探します。以下の例ではtemperature, periodを見つけることができました。
必須のdescriptorの内、自動で見つけられなかったものをマニュアルで追記します。
以上の流れで自然言語から機械可読な形にプロトコルを変形します。
実データを扱う上での注意
- プロトコルのファイル形式はtxt, tex, doc, pdfなど多岐にわたる
- 研究室間で意味が異なる場合がある
- overnight(一晩)の定義は12時間だったり18時間だったり、特に決まってないことも。。。
- 生命科学のentityは別名を多く持つ
- serotoninは5-hydroxytryptamineや5-HTと表記される(ChEBI)
- ChEBIというオントロジーで一意なURIが割り振られているのでそれを参照する
- 必要な情報が抜けていることがある
今後の展望
EXTRACT2では自然言語で書かれたプロトコルからaction, descriptor, relationの形を出力できました。
しかし、時系列を考慮しておらずまだまだ十分ではありません。
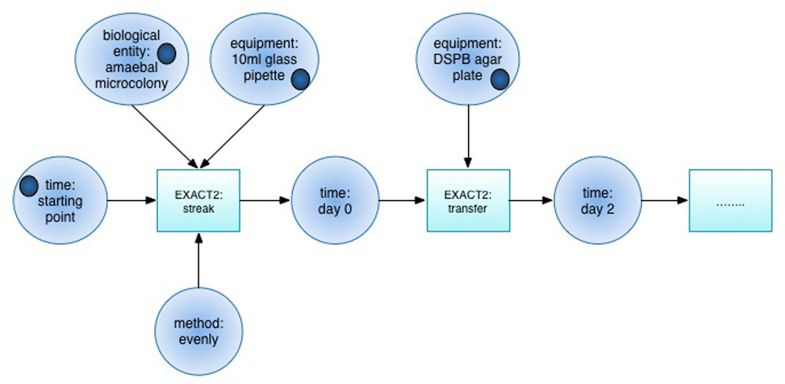
著者らはより適切な表現方法のためにペトリネットを導入しようとしています。
こんな感じで全体の流れが表現できるとのことです。
ここまで実現できたらシミュレーションにも使えるかも、だそうです。
おまけ
wet labプロトコルをアノテーションした以下の論文を紹介していただきました。
アノテーションスキームにEXACT2をベースにしたものを使っているみたいです。
僕はin Vivoに特化したプロトコルの解析ツールを開発しているので同様にマイナーチェンジして適用してみます。
プロトコルに不備があると後続の研究者のリソースが浪費されてしまいます。
本当は初めから機械可読な形でかつNULL不許可なフォーマットがあると良いんですが。
Discussionでプロトコルの不備が多くの研究者の時間とお金を浪費させた例として小保方さんの名前があげられていました。
もちろん、彼女個人を責めることで解決する問題ではなく業界のスタンダードがいけていないって話なんですが。
当時もメディアは個人あるいは研究室を非難していましたが、こういった本質的な論点に触れて欲しいものです。