マウスの系統表現③系統リスト
前回の正規表現による抽出では、情報抽出段階では問題ないが検索段階に難あり
階層構造がある
例えばC57BLの下にはいくつかの亜系統が存在する
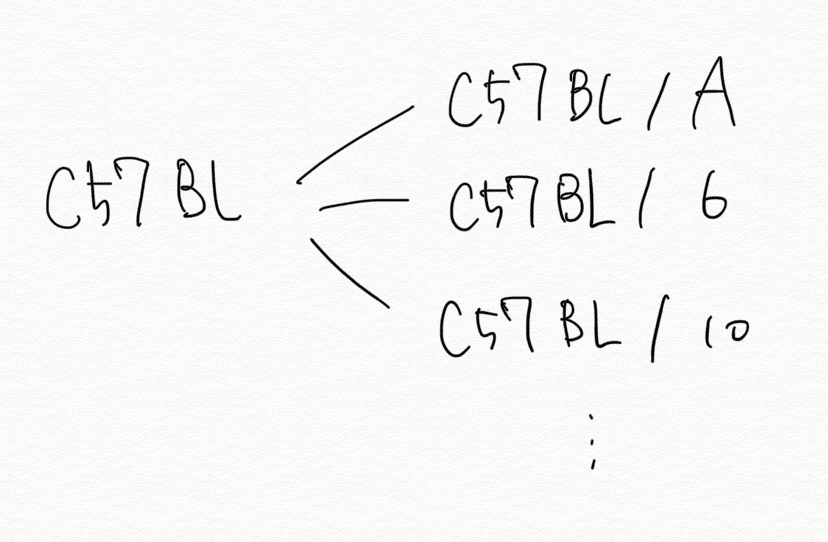
C57BLで検索した場合、それ以下の亜系統でタグ付けされたものも引っ張って来る必要がある
目次
マウス近交系の観点で論文分析
Laboratory Mice and Ratsにて分析結果が公開されていました。
ほとんどC57BL, BALB/c
PubMed論文のMeSHタグから計測したようだ
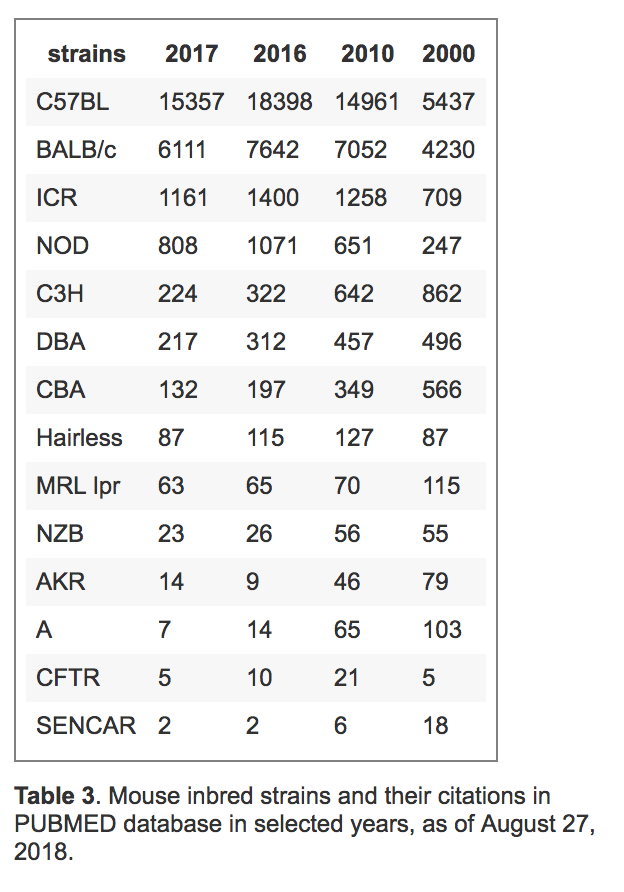
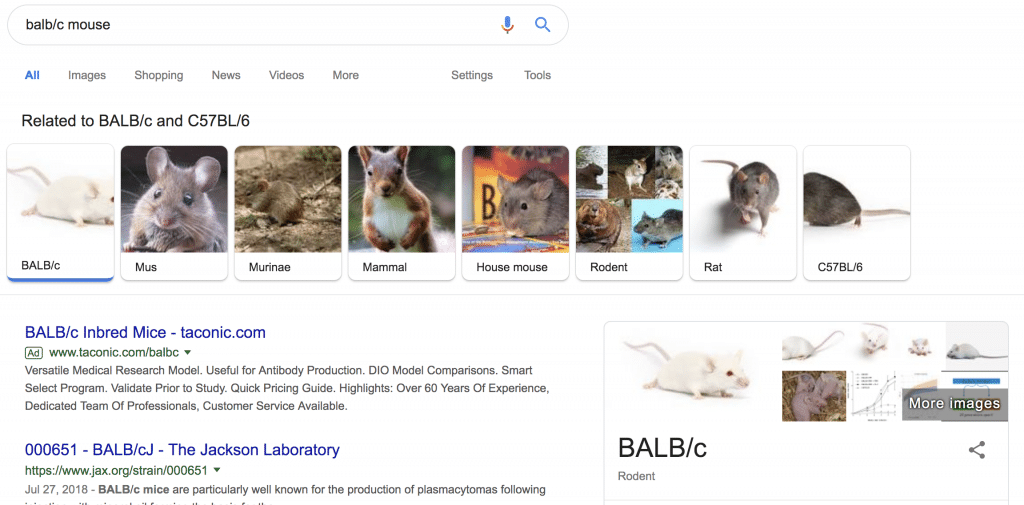
あれ、FVBランクに入らないの?と気になってGoogleのヒット数を比較した
C57BLが707万件、FVBが43万件、BALB/cが737万件、
確かにざっくり1/20、多いとは言えないかも。でも無視できる量じゃない
次項でMeSHタグに対しての指摘を述べる
MeSHによる分類
MeSHタグでマウスの近交系のリストを調べてみる
実験動物として見るか、純粋にマウスという生物種として見るかで多少異なるが、15種類くらい
つまりPubMed内にてマウスの区分は実質これくらいの粒度で行われている

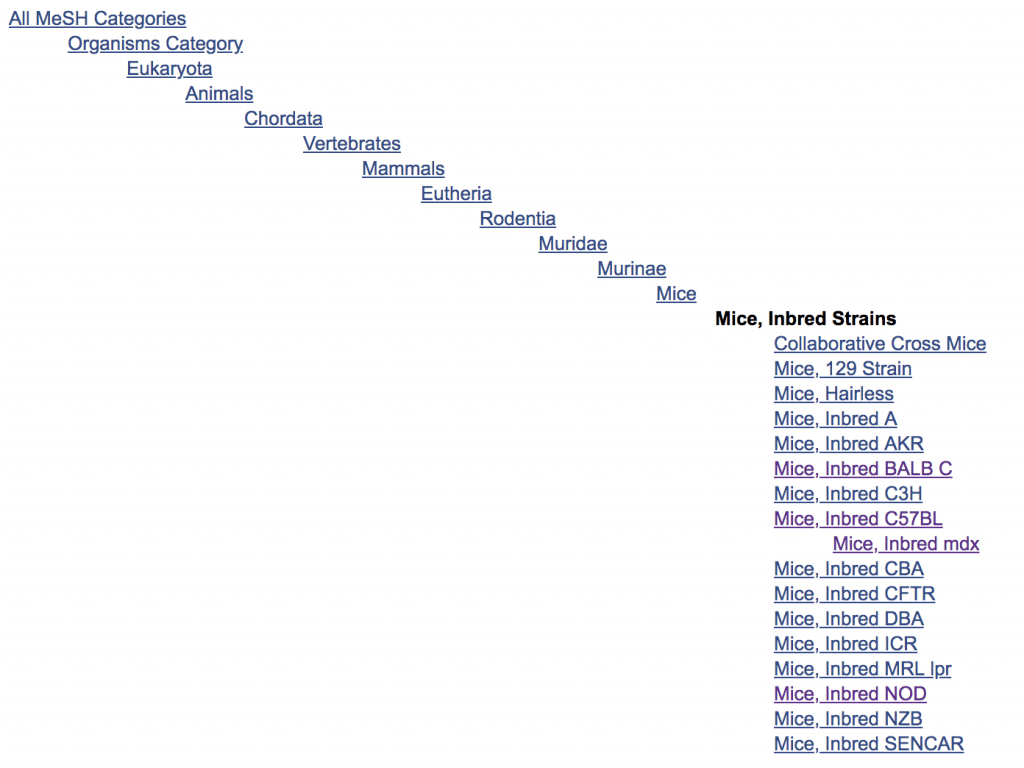
確かに僕も研究してた時、そんなにマウスの系統を知っていたわけではない
精々、Balb/c, C57BL/6, FVBぐらいをマウスの種類として認識していた
さて既にFVBがMeSHタグがない、PubMedのタグ付けが不十分なのがわかるだろう
FVBマウスの記載がある論文を開いてみる
Mice, Inbred Strainsというタグがつけられている。なんとざっくりな!

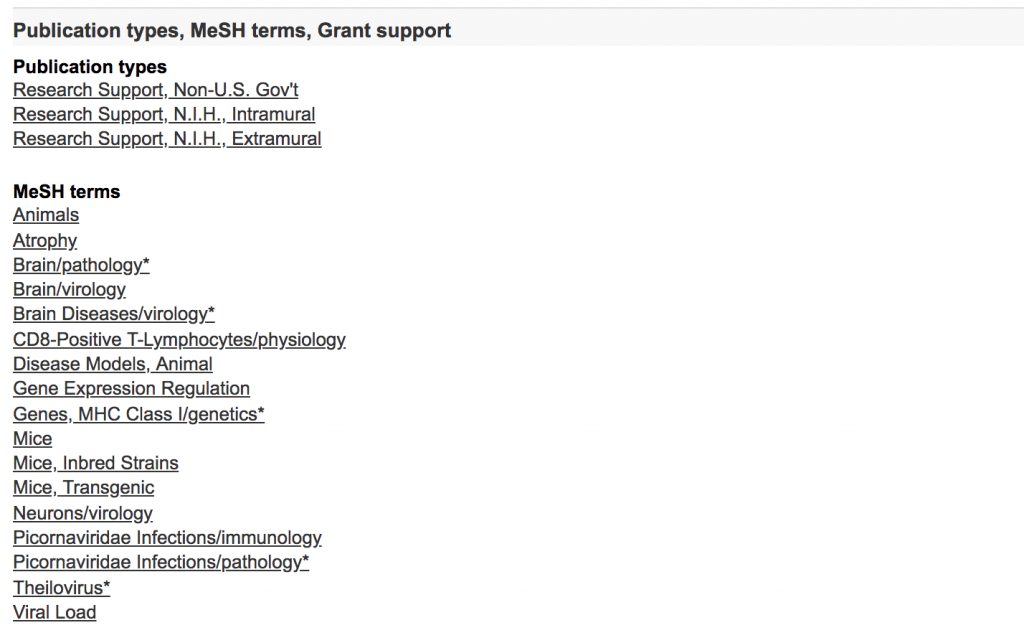
これが問題の1点目、網羅性
次に2点目の問題、粒度について実際の論文を見る
C57BLのMeSHタグが貼られている論文を探す

本文を見るとC57BL/6と記載されている
この他にもう一箇所C57BL/6と記載があり、C57BLとだけ書いてある場所はなかった
論文内では亜系統まで記載があるのに、実際にはその一段階上の粒度のタグが貼られている
結構大事な情報が落ちてしまっている

僕が開発しているシステムでは流石にこの荒さ、網羅性では使い物にならない
そこで自分でアノテーションする必要がある、というかできたらPubMed改修して欲しい
MGI
MGIでは主要な近交系426種と、それらに対して90種の亜系統が登録されている

別名もいくつか例が挙げられている

ガン関連でよく出て来るNOD SCIDは上のリストにはなかった
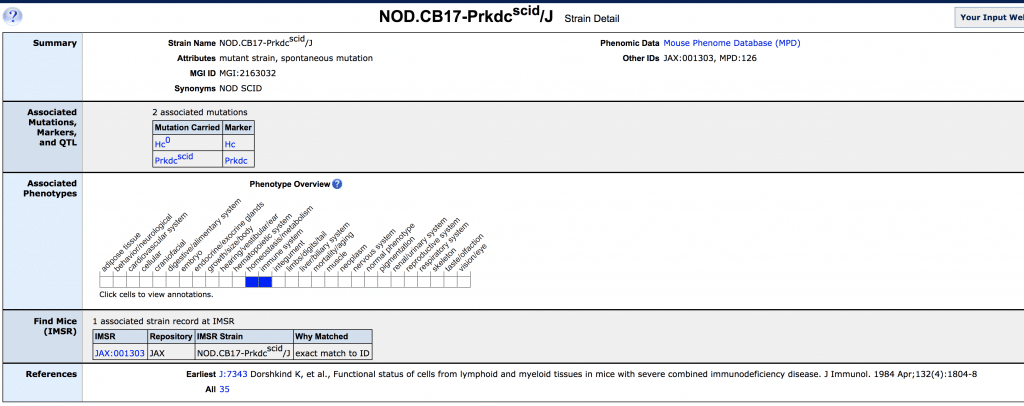
近交系の全リストには2765種が記載されているが、今度はラボコードまで書かれている(C57BL/6, BALB/cが存在しない)
横に別名が書かれているのが嬉しい

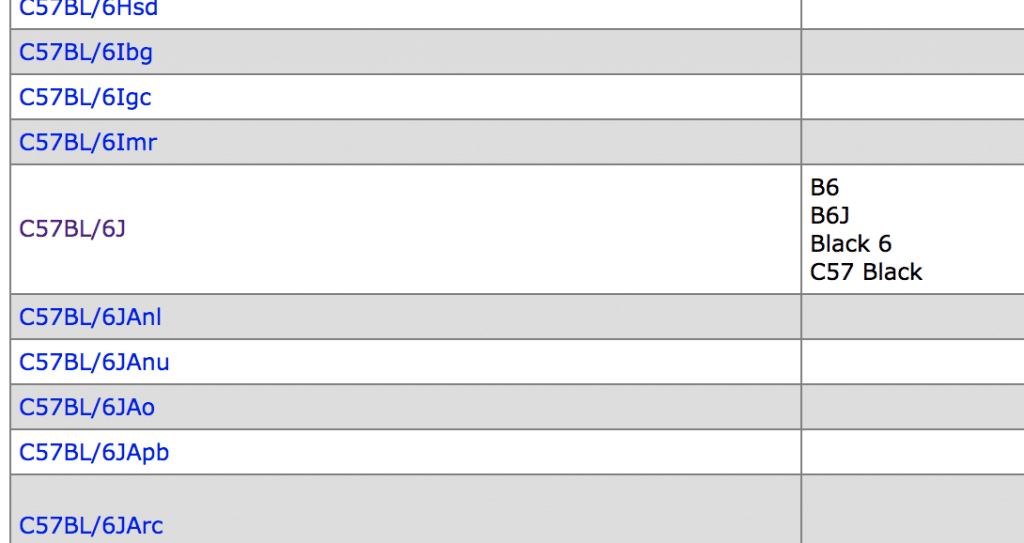
さらに範囲を広げてAll strainsには50,000強が登録されているが、どう考えてもこんなに使わない
このリストにはピュアなC57BL/6も含まれていた
選択肢は
・近交系426種 + 亜系統90種 + 別名
・近交系全2,765種 + 別名 = 3,397種
・系統全51,761種 + 別名 = 75,790種
3つ目の全種を使いたい
ただしノイズが引っかかると思うので
文字数制限や正規表現(前後に境界条件)などを考えないといけないです
抽出①マウス系統名
試しに75,790種の系統名を全て使ってみました。
全部re.escape()した後にリストに突っ込んで、パイプでjoin()してORで引っかかる正規表現を作る。
①1, 2文字が引っかかってくる
論文内の1文字、ないし2文字を大量に誤検出してしまう。
最低文字数を設定する。リストから2文字未満の名前は削除。
名前のリストは75,790種から75,505種に。
色々分析できなさすぎるので「近交系426種 + 亜系統90種 + 別名」のリストを使って以下を検証。
②固有名詞以外の単語の一部が引っかかる
適当な論文10本に対する結果(小文字にして集計)
{'pera': 19, 'star': 8, 'balb/c': 3, 'c57bl/6': 1, 'nmri': 1, 'ites': 2, 'ssin': 1}
pera, nmri, ites, ssinとか怪しいなあ。絶対マウスの系統名じゃない。聞いたことないし。
最初に読んだ論文の分析結果から考えて、BALB/c, C57BL/6より多く出てくるのはあり得ないだろう
starは一般名詞が引っかかってしまったかな
4文字以下も誤検出が多そう。しかし除去はできない(NOD, FVBのように3文字でよく使われるマウスがあるのでそれほど文字数制限を厳しくできない)
ので正規表現で前後に境界条件を与える。
うん。ちょっとまし。
{'balb/c': 3, 'c57bl/6': 1}
③全リストを利用
改めてでかいリストを使って検出してみる。時間がかかる。
加えて、なるべく長いのを見つけてほしい(C57BLよりC57BL/6で引っ掛けたい)ので文字数の降順でリストをsort()。
ノイズ多すぎ問題。
{'l line': 19, 'blast': 5, 'large': 3, 'small': 6, 'light': 8, 'caspase 3': 1, 'caspase 9': 1, 'balb/c': 3, 'fluoro': 2, 'c57bl/6': 1, 'no-2': 2, 'carrie': 3, 'plastic': 1, 'om/w': 1}
例えばcaspase 3がB6.129P2-Casp3<tm1Mak>/Cnbcマウスの別名として登録されてたのが原因。いやー、遺伝子改変マウスを改変対象の名前で呼ばんでくれーい。わけわかんなくなっちゃう。
一旦全リストの利用は保留だなあ。
論文10本に検索かけるのに3分もかかっちゃうし。
④簡易版だけ
簡易版だけで100本検索。4秒。結果はこんなもん。
{'balb/c': 27, 'c57bl/6': 24, 'c57bl': 1, 'naked': 1}
このリストに表記揺れをsynonymsとして追加していこう。
簡易版の系統名に一致する系統全種の別名とMGIを引っ張ってリストを補完。
改めて100本に対して検索をかけた
FVB/n, CF-1, Swissが引っかかりました。
{'balb/c': 27, 'c57bl/6': 24, 'c57bl': 1, 'fvb/n': 1, 'naked': 1, 'cf-1': 1, 'swiss': 5}
とりあえずマウスの系統名としてはこのリストに載っているものだけを対象にします。
抽出②性別、週齢
マウスの属性として性別と週齢を取得します
(遺伝子に関しては難しそうなので保留)
①性別
male|femaleだけでOK
②週齢
適当に正規表現を噛ませる
num = r'\d+'
add_num = '(' + ' ?( |to|,|~|-) ?' + num + ')*'
nums = r'\b' + num + add_num
ages = nums + '( |-)?(days?|weeks?|months?)' + '(( |-)(old|of age))?'
だいたい取れたと思われる
アノテーション
正規表現で取得した系統名、性別、日/週/年齢表現をアノテーションする
bratで扱えるように適当にインデックスとかをre.start()で取ったりしてテキストに応じた.annファイルを作成
(あ、ちなみにin vitro弾きたいので’xeno’が含まれない論文は弾きましたxenograft=異種移植が含まれてたらまずだいたいin vivo実験やってるはずという考え)
100本ぐらいちゃちゃっと処理した
眺めり
いい感じ一号

二号
悪い感じ一号:マウスの週齢表現じゃないただの日数にアノテーション
まぁそりゃそうだ、系統名ラベルの位置情報でage, sexはおけだめ判定してあげる必要あり

悪い感じ2号:NODが取れてない

原因、NODは3文字なので前後に境界条件が付いてしまった
記号は境界扱いだと勘違いしてた
pattern = '\b' + 'NOD' + '\b'
意図してたのは\Wでした([^a-zA-Z0-9_])、文字と数字以外
つまり空白とかカッコやカンマなんかを認識
pattern = '\W' + 'NOD' + '\W'
そっか、ドットとスペースが入る。。。

正規表現でマッチする条件には見るけど、結果には渡さない
後読みアサーション(?<=…)と先読みアサーション(?=…)を使う、苦手
こういうのパッと出せたら素敵
pattern = '(?<=\W)' + 'NOD' + '(?=\W)'

オッケー。一歩一歩頑張ろう。
とりあえずちょっと見たけど、だいぶルールを追加する必要ありそう。
あとで読めなくならないようにいい感じにコード書かないと
次回はアノテーションした100件を眺めてルール修正、
strainタグの位置との関係からsex, ageタグで余分なものを除去
そんな感じ