Biopythonを使ってPMCから論文取得
おさらい
- PubMed: 生命医学文書のインデックス
- PMC: 生命医学文書フルテキストのアーカイブ
- ClinicalTrials.gov: 臨床試験のデータベース
PubMedにはMeSH termやPublication Typeなどのメタデータが付与されていますが、テキストはAbstractしか入手できません。PMCはfull textが取得できますが、PubMedのように複雑な検索ができません。
PubMedで特定の条件に合致する論文を検索、PMCからfull textを入手する一連の流れをまとめました。
対象
課題をRandomized Controlled Trialのテキストを探すことに設定します。
filed Publication TypeにRandomized Controlled Trialを指定して検索します。49万件ヒットしました。
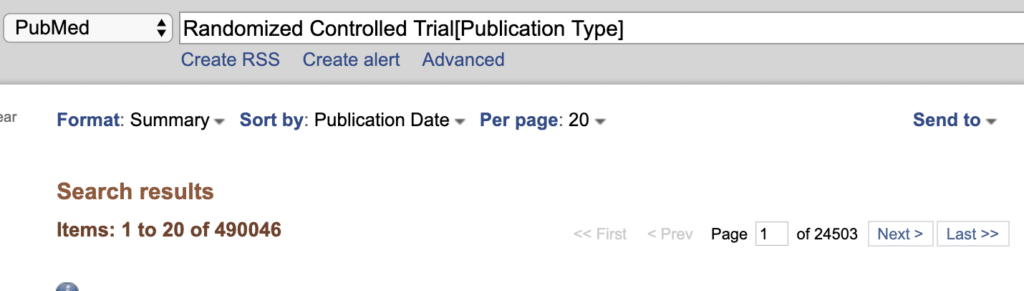
まずはこれらの書誌情報やAbstractを機械的に取得することを目指します。
色々使うので用語を整理します
- Entrez Programming Utilities (E-utilities): NCBIのデータベースを操作するためのプログラムの総称
- ESearch: 検索機能
- EFetch: ダウンロード機能
- BioPython: Entrezのラッパーとして使えるPythonライブラリ
Biopythonインストール
pipでインストール可能です。
pip install biopython
PMIDを取得
ESearchを使ってまずはダウンロードしたい論文のPMID (PubMed ID)を取得。
from tqdm import tqdm
from Bio import Entrez
Entrez.email = YOUR_EMAIL
pmids = []
term = 'Randomized+Controlled+Trial[pt]'
db='pubmed'
retmax = 1000
handle = Entrez.esearch(db='pubmed',term='Randomized+Controlled+Trial[pt]')
record = Entrez.read(handle)
count = int(record['Count'])
for retstart in tqdm(range(0, count, retmax)):
handle = Entrez.esearch(db=db, term=term, retmax=retmax, retstart=retstart)
record = Entrez.read(handle)
pmids.extend(record['IdList'])
スペースを挟む連語を検索ワードに使うときは+で繋ぎましょう。[pt]はpublication typeです。はじめからfree full textのものだけを取得したければterm = 'Randomized+Controlled+Trial[pt] AND free+full+text[sb]'とします。細かいオプションなどは公式を確認してください。
RCTは49万件、そのうちfree full textにアクセス可能なものは13万件あります。
論文を取得
EFetchを使って前項で取得したPMIDの論文を取得します。
retmax = 1000
for i in tqdm(range(0, count, retmax)):
handle = Entrez.efetch(db='pubmed', id=pmids[i:i+retmax], retmode='xml')
text = handle.read()
with open('{}-{}.xml'.format(i, i+retmax), 'w') as f:
f.write(text)
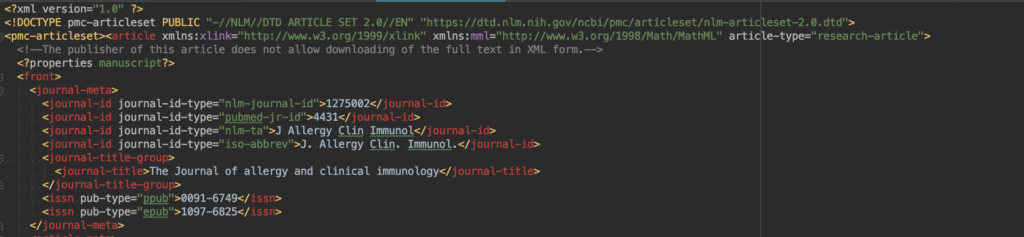
こんな感じで書誌情報とともにAbstractを取得できます。

詳細は公式。
EntrezではEFetchに200より多いIDを渡す場合はPOSTメソッドを使うように言われています。Biopythonでは自動でよしなにしてくれます。ちなみに10,000件一気に取ろうとしたら1リクエスト2分半かかりました。
PMIDに対応するPMCIDを取得
PMCにフルテキストがある論文はLinkOutにPMCへのリンクがあります。

ただしfree full textの条件を入れたとしても、必ずしもPMCIDが紐づいているとは限りません。以下一例。

先ほど取得したPubMedの情報からPMCIDを取得することができますXMLのArticleIdListに記述されています。
<ArticleIdList>
<ArticleId IdType="pubmed">31360445</ArticleId>
<ArticleId IdType="doi">10.7189/jogh.09.020402</ArticleId>
<ArticleId IdType="pii">jogh-09-020402</ArticleId>
<ArticleId IdType="pmc">PMC6657003</ArticleId>
</ArticleIdList>
ID Converter APIを叩いて確認することもできます。1リクエスト200 IDまでという点に注意します。
tool = YOUR_TOOL_NAME
email = YOUR_EMAIL
ids = []
retmax = 200
for i in tqdm(range(0, len(pmids), retmax)):
url = 'https://www.ncbi.nlm.nih.gov/pmc/utils/idconv/v1.0/?tool={}&email={}&email&ids={}&format=json'.format(tool, email, ','.join(pmids[i:i+retmax]))
r = requests.get(url)
j = json.loads(r.text)
ids.extend(j['records'])
print(sum([1 for id in ids if 'pmcid' in id))
84550
Publication Type: Randomized Controlled Trialでヒットした49万件のうち8.5万件のfull textがPMCで公開されていることがわかりました。
全文を取得
再びEFetchを使ってPMCから全文を取得します。
pmcids = [id['pmcid'] for id in ids if 'pmcid' in id]
retmax = 1000
for i in tqdm(range(0, len(pmcids), retmax)):
handle = Entrez.efetch(db='pmc', id=pmcids[i:i+retmax], rettype='full', retmode='xml')
text = handle.read()
with open('{}-{}_pmc.xml'.format(i, i+retmax), 'w') as f:
f.write(text)
先ほどのPubMedのデータとマージすればMeSHなどのメタデータと全文を合わせたものが入手できます。
だいたいこんな感じ。ジャーナルや論文自体のメタデータはPubMedのデータとダブりあり。
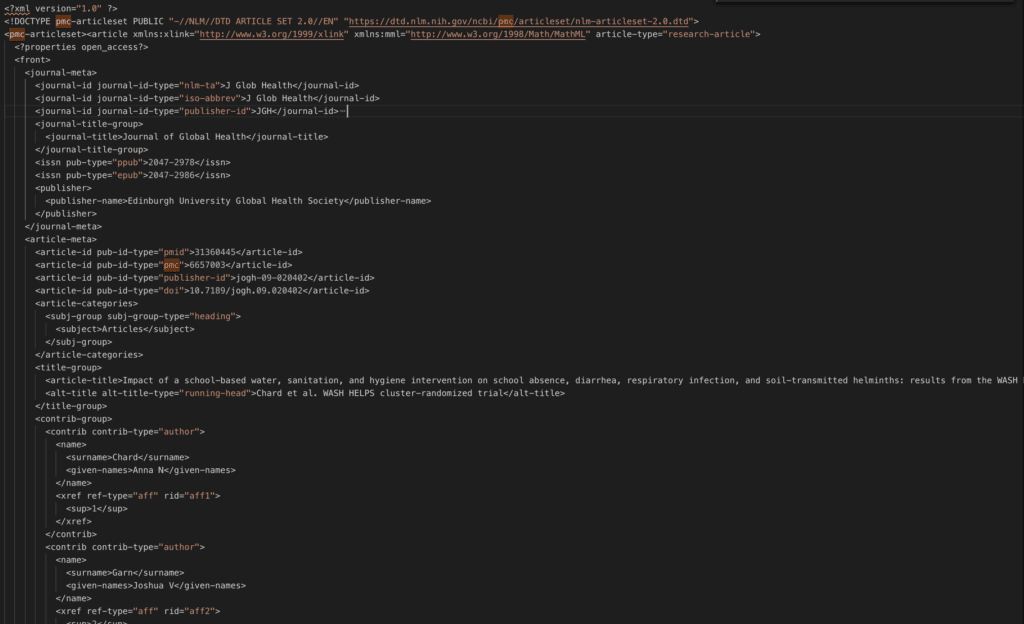
ただし出版社によってはfull textのダウンロードを禁じている場合があります。その場合は4行目に”The publisher of this article does not allow downloading of the full text in XML form.”とコメントが入ります。