gitでアノテーション管理
論文解析をズーーーっとやってます。
どんな種類のタグをつけるのか、どこまでの粒度でつけるのか、
ルールを作っては壊し、直しては白紙に戻し、
最近ようやく方向性が決まりました
目次
解決したい苦痛
論文の実験条件のまとめ、比較は作業であり面倒
つまらないのに時間がかかる
どれだけ頑張っても網羅することはできない
やりたいこと
論文から実験条件を抜き出す
そのデータを使って適切な実験条件を提示する
第一段階として成し遂げたいこと
腫瘍の異種移植実験に関する論文からその実験条件を取得する
具体的には
- mouseのstrain, sex, age
- cancerのcell-line, number, route
- reagent(薬)のname, number, route
新しい方針
第一段階が成功したかどうか確認するプロセス
<h3>タグにmouse, mice, xeno, implantが含まれるかでフィルタリング
- 含まれていたら見出しと本文を取得→次のステップに利用する
- それ以外は破棄→使わない
- 一つも見出しが引っかからないファイルは使わない
- 結果:cancerで検索したnature c, scientific rの20%がフィルタリングを通過:8610 files → 1729 files
gold, train, testに分割、以下のディレクトリに配置(gold, testは同一ファイル)
- man/ gold, test: 50, 50
- auto/ gold, train, test: 50, 1629, 50
- goldはregex taggerの訓練用
- trainはdl taggerの訓練用
- testはdl taggerの評価用
まずはmouseに関するタガーを作る
regex tagger:正規表現タガー
- auto/ gold, testの100件に対してリストと正規表現でタグ付け
- man/ gold, testの100件に対して、autoの同ファイルを元にmouseタグを手作業で全部修正
- regex taggerをauto/goldに適用man/goldと完全に一致するまでルールを改修する(regex tagger精度100%を目指す)
dl tagger:深層学習タガー
- ある程度確からしいregex taggerでauto/trainをタギング
- auto/trainを使ってdl taggerを学習
- auto/testにdl taggerを適用、man/testと比較し精度を出す
まずはmouseのタグで上記工程を実施し、cancer, reagentで同様に繰り返す
reagentはnameだけlampleの学習済みモデルを使いたい
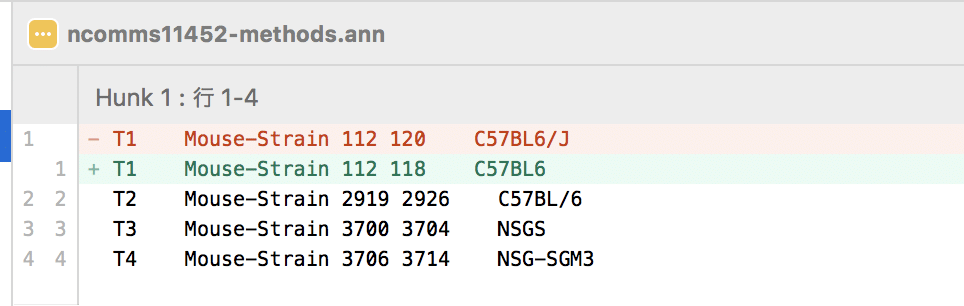
gitを使って楽に差分を確認
まあでもregex tagger変更加えると、意図しないルールの変更でチェック済みのファイルがうまくタグ付けされているか保証がありません
たかが50件程度ですが、この確認はしんどい
ってことで、ディレクトリをSource Treeで管理することで、diffが簡単に取れるようになりました
便利!