PubMed分析
PubMedの論文タグを集計して、疾病ごとの論文数推移を見ました。
家畜伝染病・代謝疾患・内分泌系疾患などが伸びていました。
目次
PubMedとは
一言で言えば生命科学論文の検索エンジンです。
少し詳しく言うと、
アメリカの国立衛生研究所(NIH)が国立医学図書館(NLM)という医学図書館を持っています。
NLMの一部門に国立生物工学情報センター(NCBI)と言うものがあって、データベースの構築などを担っています。
PubMedはNLMがこれまで収集してきた生命科学領域の論文情報に対する検索エンジンです。
要するに、キーワード入力すれば古今東西のジャーナルからマッチする論文を探してくれるやつです。
MeSH (Medical Subject Headings)とは
生命科学の論文や書籍をカテゴライズするシソーラスです。
NLMにより定義されており、2018年版では28939のdescriptor(識別子)が登録されています。
現在使われているMeSH descriptorsはこちらから入手可能です。
MeSHは木構造で構成されています。
まずAnatomy, Organismsなどの16の大分類を有し、その下により詳細な分類が続きます。
Anatomyの下には解剖学的観点での分類が続きます。
Body Regions(体の部位)やCells(細胞)といったようにまた別の観点での区分が続きます。
(Body Regionsの下には足とか腕とか、Cellsの下には赤血球とか白血球とか)
そしてこのような分類に対して、descriptorが紐づいています。
重要なのは多対多の関係になっていること、つまりdescriptorは複数の分類に所属しうるということです。
(descriptorは最下層だけではなく、途中の枝にも紐づいています)
例えばErythrocytes(赤血球)というdescriptorは以下の3つのtree numberに紐づけられています。
下の画像を見るとErythrocytesは細胞の種類による分類と、血液の構成成分による分類に使われていることがわかりました。
PubMedにおけるMeSHの利用
PubMedでは複数のMeSHが論文に割り振られています。
これを集計すれば簡単に分析できるかな、と思ったので実際どのように使われているか確認しました。
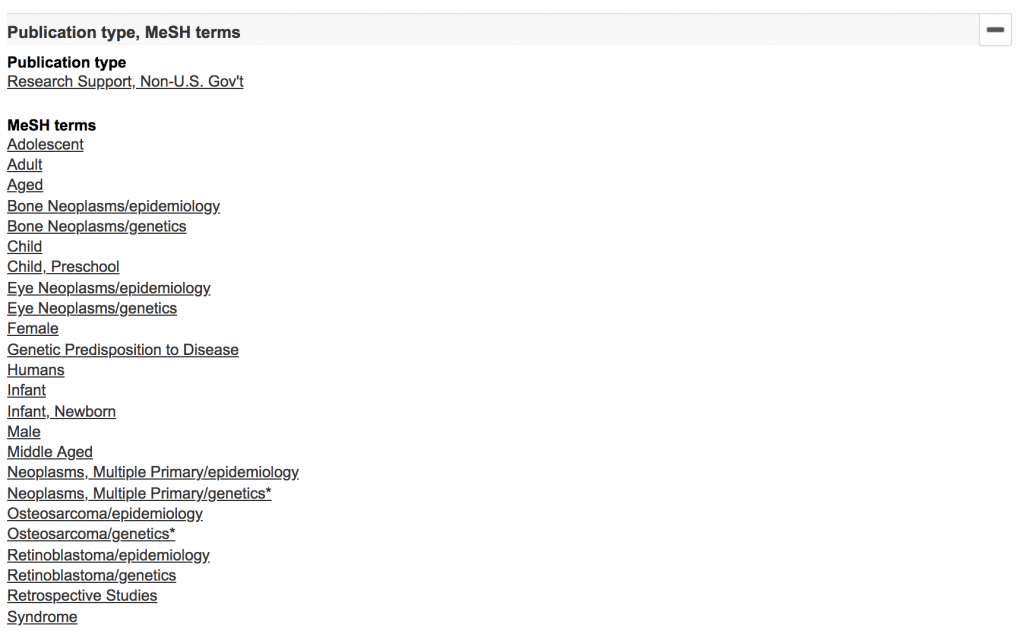
例として以下の論文「骨肉腫患者における多発性原発悪性腫瘍。骨肉腫に関連する癌症候群の骨肉腫サブタイプの発生率と予測値。」(Google翻訳)を使って解説します。
(めんどくさいのでこの項はスキップしても全体の理解には差し支えないと思います)
出展: PubMed
ざっくり読むと、骨肉腫(osteosarcoma)患者は別の悪性腫瘍(malignancy)を併発していることがあるのでそれを分析したって感じです。
ネタバレをすると、osteosarcomaのサブタイプを分析することで併発する悪性腫瘍の傾向がわかる、かも、というものでした(流し読みなので多分)。
下に進むと以下に示す、「Adolescent」から「Syndrome」までの24のMeSHでラベリングされていることがわかりました。
出展: PubMed
Humansとあるので人間のデータを使ったと想像できます。
さらにInfant, Child, Adolescent, Adult, Agedから年齢、Female, Maleから性別の2つの観点で分析されたことが推察できます。
実際に論文のFigを見ると、タグから推測できるような形をしていました。
またOsteosarcomaは今回の着眼点、Bone Neoplasmaはその上位概念です。
Retinoblastomaは網膜芽細胞腫のことで、Eye Neoplasmaはその上位概念です。
以下はICD-10という疾病の分類の国際基準です。C00-C97にはMalignant neoplasms(悪性新生物)が列挙されています。
先ほどの2つもこの範囲に収められています。
ざっと読んだ限りでは、併発する疾患の一つとしてRetinoblastomaが取り上げられいたのでMeSHが割り振られたようです。
他に多くの腫瘍が挙げられている中で(特にCarcinomaなどは並列で書かれているのに)Retinoblastomaだけ特別にタグがあった理由は不明、Abstructに記載があったから?
読み逃しただけで強い相関が見られたのかもしれないです。
と、論文の中までちょっと読んでみましたが、設定されたMeSHから大まかに論文をカテゴライズすることができそうだとわかりました。
(あくまで大まかにですが)
論文のMeSH情報を取得できないか探します。
Entrez & E-Utilitiesとは
EntrezはPubMed同様NCBIに運営されるシステムです。
EntrezはPubMedを含め、タンパクや遺伝子単位での情報を保存したデータベースです。
細かい説明は公式サイトを引用します。
Entrez is a molecular biology database system that provides integrated access to nucleotide and protein sequence data, gene-centered and genomic mapping information, 3D structure data, PubMed MEDLINE, and more. The system is produced by the National Center for Biotechnology Information (NCBI) and is available via the Internet.
出展: Entrez
EntrezはE-UtilitiesというAPIを介して利用します。
E-Utilitiesの簡単な説明は以下の動画を参考にしてください。
詳しい説明はこちらを参考にしてください。
eutilityは全部で9種類があります。
基本的な書誌データを取得するesummaryやMeSHやabstructまで取得するefetchなど。
以下のようにfcgiの前を変更することで取得するデータを変更できます。
- https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi
- https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi
まず各種パラメータを紹介します。
id(論文ID)
idパラメータに論文IDを指定することで特定の論文のメタデータ(書誌データ)や本文を取得します。
著者情報やタイトル、ページ数などの情報がわかりました。
- https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi?db=pmc&id=3539452&retmode=json

term
キーワード検索ができます。
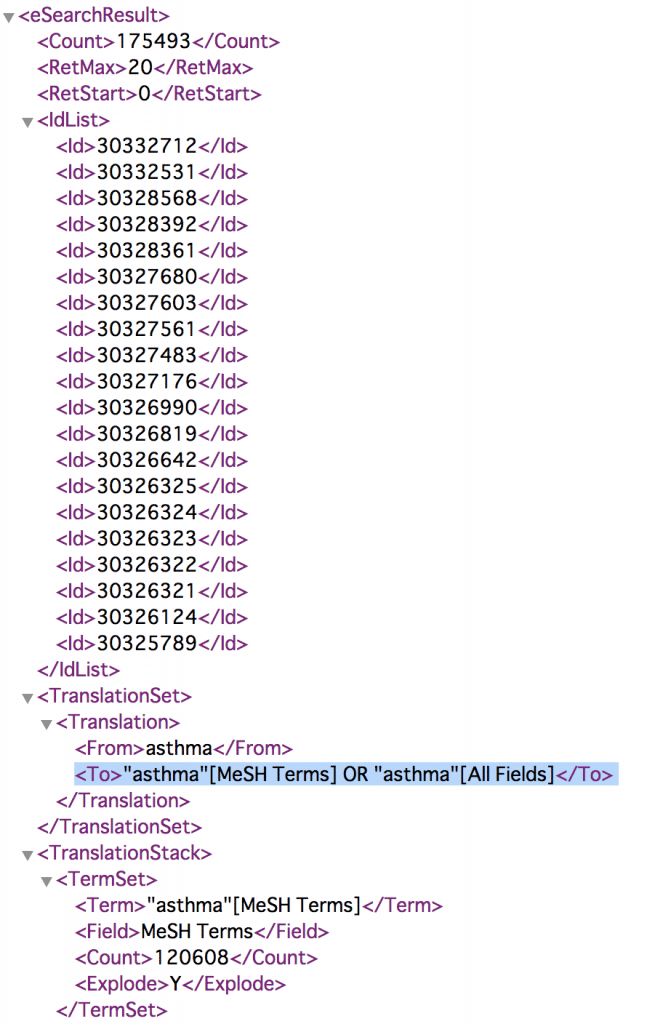
例えば「term=asthma」とすれば
- https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=asthma
以下のようにMeSHと全てのfieldに対してasthmaが検索されていることがわかります。
asthma[mesh]とすることでMeSHからだけの検索とできるみたいです。
- https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=asthma[mesh]
(このルールに関して明確に記述されているページを見つけることができませんでした。いくつかfieldにmeshを設定して返ってきた論文idを確かめましたが、その範囲では該当論文のページでちゃんとMeSHが設定されていました。)
field
先ほどのasthma[mesh]の書き方(上)はfieldパラメータを使って下のように記述できます。
- https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=asthma[mesh]
- https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=asthma&field=mesh
retmax
取得数の制限です。デフォルトでは20件までしか取得できないのでこの数字をいじることで増やします。
最大でも100,000なので気をつけましょう。
取得できなかった分についてはretstartを使ってペジネーションのように取得します。
retstart
取得の開始インデックスを指定します。
mindate, maxdate
これらのパラメータを設定することで日付の制限を設けることができます。
日付のフォーマットはYYYY, YYYY/MM, YYYY/MM/DDの3種類が使えます。
E-Utilitiesで疾病ごと論文数の経年推移を確認する
ようやく本題です。やりたいのは疾病という観点で論文数の推移を確認することです。
イメージでいうと、発行年と疾病でグルーピングして数を取る感じ。
検索対象の疾病を定義する
MeSHデータベースを叩いて子要素をリストで取得する方法が見つからなかったので、HPから直接スクレイピングしました。
Disease直下の26要素のMeSH termを取得。
上で取得したMeSH termを使って以下のエンドポイントを叩きます。
- https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=<MeSH term>[mesh]&mindate=<YYYY>&maxdate=<YYYY>
XMLが取得できるので、BeautifulSoupなりでパースして、Countの部分(条件を満たす論文数)を取得すればOKです。
上記のプロセスをコードに落とし込んで各MeSH term、年次の組み合わせでCountを取得。
ヒートマップにしてみました。
2018年が少ないのは当然として、2017年も全ては登録されてなさそうです(増加し続けていたのに2017年だけ少ないので)。

ぱっと見、目立って論文数が多い領域はこんな感じですが、
- Neoplasms:新生物(がんとか)
- Nervouse System Diseases:神経系疾患(パーキンソンとか)
- Cardiovascular Diseases:循環器系疾患(心不全とか)
- Pathological Conditions, Signs and Symptoms:病的状態、兆候、症状など(低体温など)
4つ目は区切り方が異なるような気がする。モヤモヤ。
そもそも下層に含まれる要素の数がわからないので論文数での比較はフェアじゃないかも。
各項目の2000年時点の論文数を1として相対値で表してみました。
こうして見ると、以下の項目が2000年と比較して増えてきているようです(ちゃんと計算したらNeoplasmsは上昇率4位でした)
- Neoplasms:新生物(がんとか)
- Nutritional and Metabolic Diseases:栄養病、代謝疾患(糖尿病など)
- Animal Diseases:家畜伝染病(鳥インフルエンザなど)

ここまでヒートマップで視覚的にざっくり確認しただけなのでしっかり数字を出してみます。
2000年と2016年を比較して、論文数の上昇率トップ3とワースト3を取得しました(%は2000年比)。
トップ3
- Animal Diseases:家畜伝染病(鳥インフルエンザなど):283%
- Nutritional and Metabolic Diseases:栄養病、代謝疾患(糖尿病など):234%
- Endocrine System Diseases:内分泌系疾患(バセドウ病など):212%
ワースト3
- Disorders of Environmental Origin:環境由来疾患(化学物質過敏症など):60%
- Occupational Diseases:職業病(塵肺など):116%
- Hemic and Lymphatic Diseases:血液疾患、リンパ性疾患(血液凝固障害など):147%
繰り返しになりますが、疾病の中には複数の疾病カテゴリに所属するものもあります。
例えば、糖尿病は代謝疾患と内分泌系疾患の両方に含まれます。
こんな感じ。
論文数自体は2000年比、226%に増加。16年で2倍以上論文投稿数が増えているということになります。
トップ3とワースト3を論文数自体の伸びと比較しました。

トップ2だけが平均を上回る結果になったのはなんか変な感じします。
おまけ
論文数が増えているのに比べて各疾病カテゴリに割り振られる論文数の上昇率が低いのは、一つの論文に対して割り振られるMeSHの数が減っている(判断基準)が変わっているのかもしれません。
もしくはMeSH自体毎年更新されているので2000年から2016年のタイミングで結構変化があったのが原因かも。
今回の分析で定量的な部分は信用ならないけど、トップ3、ワースト3を見るとなんとなく雰囲気は伝わる。
環境とか職場とかの汚染による疾病が減ってきた(絶対数にしろ関心にしろ)というのは自分の感覚とも一致します。
代わりに代謝疾患とか内分泌系疾患(自律神経失調症とかが関心大きいのかなと予想)とかへの関心が増えてきた、と。
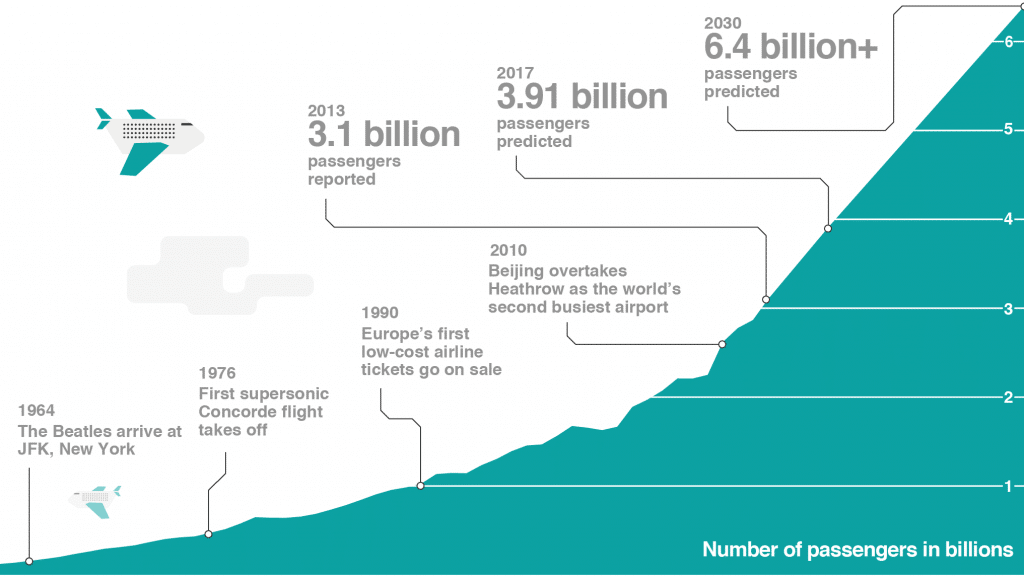
家畜伝染病がこんなに上昇率が高いとは思いませんでした。飛行機の大衆化?時系列的にタイミング一致しますかね?雑な予想。
飛行機の利用者数の推移。それほど2000年から現在にかけて急増したようには見えないです。何が原因なんだろうか。
出展: Is jet travel becoming the dirtiest way to cross the planet? |BBC








 出展:
出展: