SageMaker Ground Truthのカスタムラベリング機能
最良のテキストアノテーションサーバについて考えていました。SageMaker Ground Truthなら自由度・効率性の点で優れているとオススメされたので試してみました。躓きどころをまとめました。
Lambdaを用いた事前事後処理の自由度はかなり高そうです。ラベリングの挙動もJavaScriptを作り込めば実質どのようなものでも再現できそうです。アノテーション後のデータもS3に自動で保存されるので、後続の処理も行いやすそうでした(マニフェストファイルの形式に合わせさえすれば)。
目次
やりたいこと
私たちは生命科学論文から実験条件の情報抽出に取り組んでいます。例えばがんの移植実験に関する論文から、マウスの系統名・週齢、移植したがん細胞の株名・投与量、投与した薬剤の名称などを抽出しています。
御察しの通り週齢や投与量に関する表現は正規表現で大部分抽出できます。マウスやがん細胞の名称もあらかじめ用意した単語辞書とのマッチングである程度取得できます。
一方で薬剤名の抽出は難易度が高く、頭を悩ませていました。別の機会にご紹介するつもりですが、紆余曲折の末深層学習を用いたNERを行うことにしました。
類似のデータセット(abstractに対する薬剤名NER)と深層学習を用いた検証で10,000 docsデータがあればf1 90%近い性能を出せることは確認しています。
そこで我々の解きたいタスクに特化したデータセットを用意するモチベーションが生まれたわけです。
既存のテキストアノテーションツール比較
しばらくはbratを使っていましたが以下の要望から乗り換えを検討しています。
- 能動学習を適用してアノテーションコストを削減したい
- NERタスクに適したフォーマットで出力したい
- アノテーション中にラベルの追加・変更を行いたい
- S3などストレージサービスと連携したい
- 作成済みのタグ付けモデルを利用したい
いくつかのアノテーションツールを調べた結果、高機能(加えて高価格)なものでは能動学習、アノテーターの進捗管理などが実装されていました。
ただ、やりたいことがモリモリなので、いずれも何かが足りないという状況。スクラッチから作るしかないかなーと思ってたところ、SageMaker Ground Truthをオススメされました。
SageMaker Ground Truthとは
awsが提供するアノテーションを効率化するサービスです。
能動学習が導入されているため、効率的にアノテーションすべきデータを提示してくれます。コストと時間を削減することができるとのことです。
またMechanical Turkと連携してクラウドソーシングすることもできますし、もちろんプライベートなメンバーだけで共有することもできます。
現状デフォルトで対応しているタスクは以下の4つ。
- 文書分類(文書にラベルづけ)
- 画像分類(画像にラベルづけ)
- 物体検出(画像のある位置にバウンディングボックス)
- セマンティック・セグメンテーション(ピクセル単位のラベルづけ)
デフォルトではNERなどの系列ラベリングには対処できません。しかしカスタムタスクで自由にタスクを設定することが可能です。このカスタムの自由度がかなり高いとのことです。
今回はGround Truth custom labeling workflowの紹介記事の再現を通してGround Truthの使い方を覚えました。
SageMaker Ground Truth custom labeling workflow
custom labelingの事例はIEEEのものがaws公式ブログで紹介されています。CloudFormation, Lambda, S3, IAMへの基本的な理解が必要です。以下では次の記事に沿いながら、詰まりどころを補足しました。
Ground Truth一般の話を理解したければこちらもaws公式ブログがあるので一通り読んでみてください。
全体の構成の意図や目的は元記事に譲り、実際に手を動かす部分で初心者的に詰まったとこを未来の自分のために残します。大まかな流れはこんなとこ。
- IAMロール作成
- CloudFormationでLambdaデプロイ
- S3に入力データを記したマニュフェストファイルをアップロード、出力先のディレクトリ作成
- SageMakerでラベリングジョブ作成
IAMロール作成
CloudFormationが利用するIAMロールを作成します。ロール名はgt-custom-testとします。
「このロールを使用するサービスを選択」でCloudForamationを選択。
アクセス権限は以下のものがあれば十分です(多分FullAccessは過剰なので必要最小限の権限に変更してください)。
- LambdaFullAccess
- IAMFullAccess
CloudFormationスタック作成
今回の構成ではファイルがアノテーションされる前後にLambdaによる処理が行われます。元記事では「プレラベリングタスクおよびポストラベリングタスクの Lambda 関数のデプロイメント」と書かれています。テンプレートはGithubリポジトリにあるserver/processing/cfn-template.jsonをダウンロードして使います。
とりあえずGithubリポジトリgt-custom-workflowをクローンします。
git clone https://github.com/nitinaws/gt-custom-workflow.git
CloudFormationを開き、リージョンを米国東部 (バージニア北部) us-east-1に変更します(以降全てのリソースのリージョンを揃える必要があるので注意)。
テンプレートをアップロードでserver/processing/cfn-template.jsonを選択します。
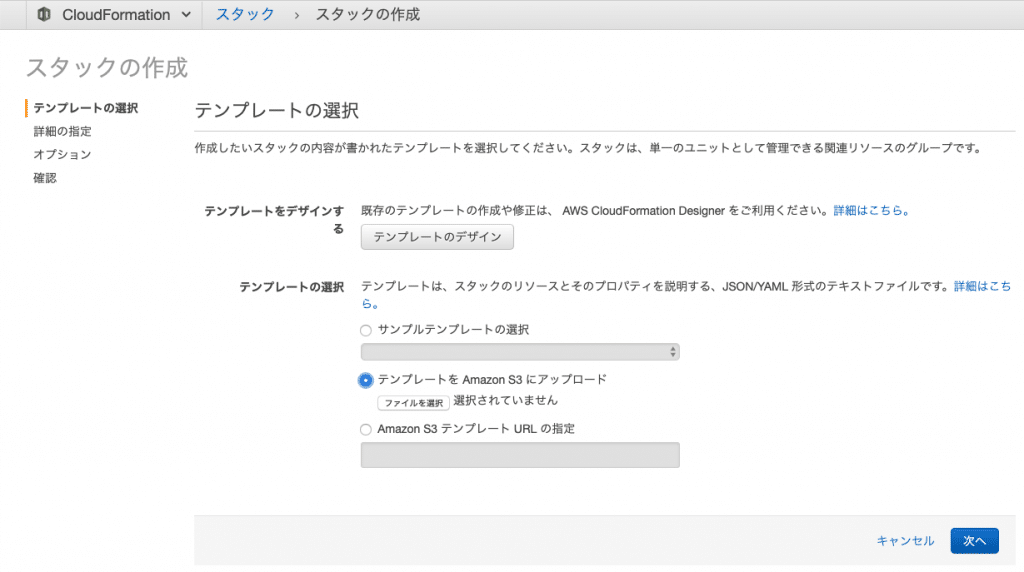
スタック名はgt-custom-testとしました。IAMロールの選択では先ほど作成したgt-custom-testを選択してください。
Lambdaコンソールを開き、バージニア北部リージョンに変更し関数を確認。pre, postのLambda関数が作成されていれば成功です。
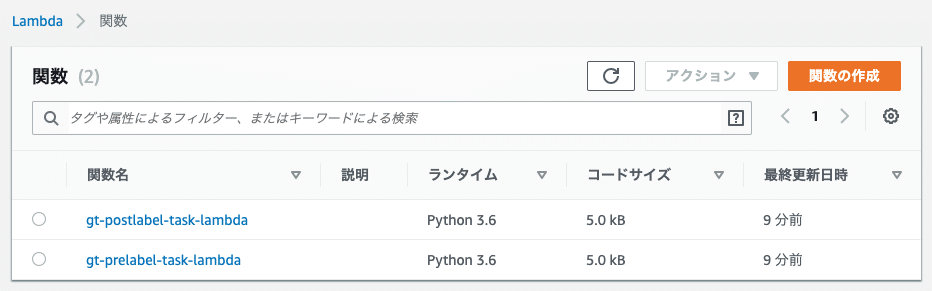
作成がうまくいっていたらCloudFormationで今作成したスタックの出力からPostLabelingLambdaIAMRoleの値をコピーしておきます。
なおスタックの作成に失敗すると自動でロールバックしてくれます。(東京リージョンで作ろうとしたり、IAMの権限漏れで何回か作り直しました。。。)
S3で入力データのマニフェストと出力先を作成
入力データの定義にはjson形式の拡張マニュフェストを用います。ここでどのファイルを読み込むか定義しています。さらにそれぞれのファイルに関するメタデータを設定できます。
入力データ(ここではaws側が用意したIEEEのabstractから抽出したテキスト)とマニフェストファイル(自分のS3にアップロード)はSageMaker Ground Truthと同じリージョンに存在しなくてはいけません。入力データのIEEE abstractはawsによって米国東部 (バージニア北部)に用意されているので全てこのリージョンに合わせる必要があるわけです。
(ブログではこれで上手くいくとのことでしたが、僕の環境では入力データを読み込むことができませんでした。後述しますがダミーデータを自分のバケットに配置すれば上手くいきます)
では米国東部 (バージニア北部) リージョンにgt-custom-testというバケットを作成します。入力データの定義としてGithubリポジトリのserver/data/manifest.jsonをアップロードします。出力先としてoutputフォルダを作成します。
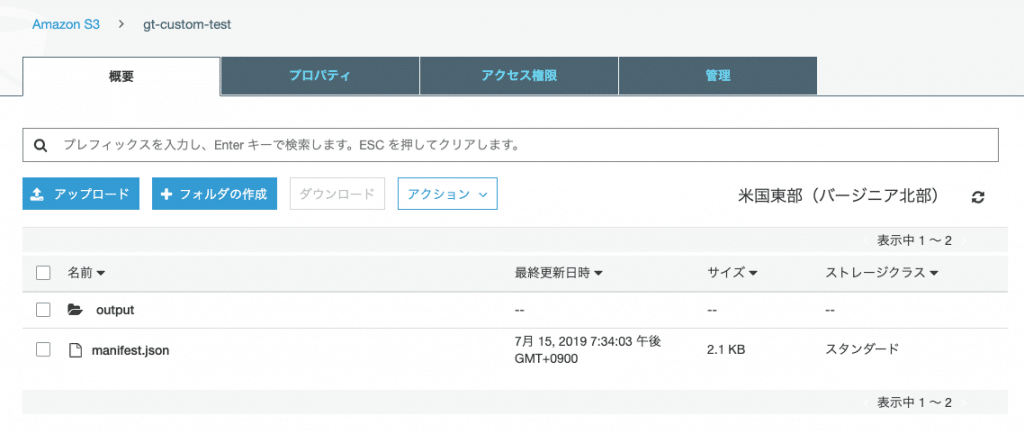
ちなみに、manifest.jsonに書かれたファイルを適当に開いてみます。
import boto3
s3 = boto3.resource('s3')
bucket = 'smgtannotation'
key = 'text/1801_00128.jpg.csv'
obj = s3.Object(bucket, key)
text = obj.get()['Body'].read().decode('utf8')
print(text)
Special Issue on the foundations of astrophysics and cosmology manuscript No.
(will be inserted by the editor)
Hubble law : measure and interpretation
Paturel Georges
Teerikorp Pekka
Baryshev Yurij
Reccivad dato Acceptod date
Abstract We have had the chance to live through fascinating revolution
in measuring the fundamnenta empirical cosmnological Hubble law. The key
...(改行省略)
ちゃんと読めますね。
なおこのテキストはAmazon Textractで論文の画像から抽出されたものだそうです。詳しい処理はserver/prep/detect_lines.pyに書いてあります。
SageMakerでラベリングジョブを作成
やっとこさSageMakerにたどり着きました。公式ブログで書かれている通り、米国東部 (バージニア北部) リージョンであることを確認してからラベリングジョブを選択。

ジョブ名はgt-custom-test、入力データセットの場所はS3のmanifest.jsonのパス、出力データセットの場所はoutputのパスを指定します。
IAMロールはCreate a new roleを選択。manifest.jsonが入っているバケットを指定します。これでこのIAMロールはS3内のファイルを扱うことができます。
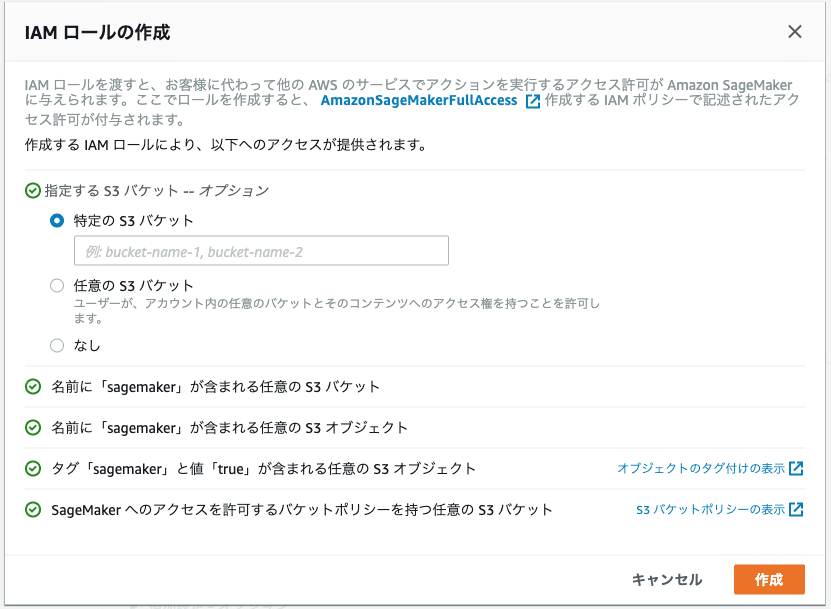
これでIAMロールが作成されましたが、今回はPost-labelingのLambda関数にS3へのアクセス権を与える必要があります。

別タブでIAMコンソールを開いて今作ったIAMロール(上画像のAmazonSageMaker-ExectionRole-xxx)を検索します。
公式ブログの「前提条件」の3に従って信頼ポリシーを書き換えます。CloudFormation作成後にメモしたPostLabelingLambdaIAMRoleの値を使いましょう。さらにインラインポリシーの追加を行います。
完了したらSageMakerのタブに戻り、タスクタイプでカスタムを選択します。
ワーカーを設定します。最低一人は招待相手としてメールアドレスを記載します。
テンプレートはカスタムを選択。Githubリポジトリのweb/public/template.htmlの中身を貼り付けます。
Pre-labeling, Post-labelingのLambdaをそれぞれ指定したら作成。
できた!
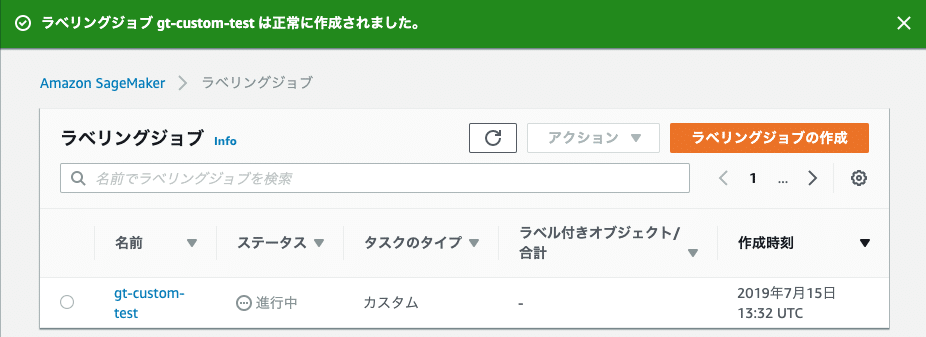
と思ったら失敗。manifest.jsonで指定したファイルの読み込みに失敗している?
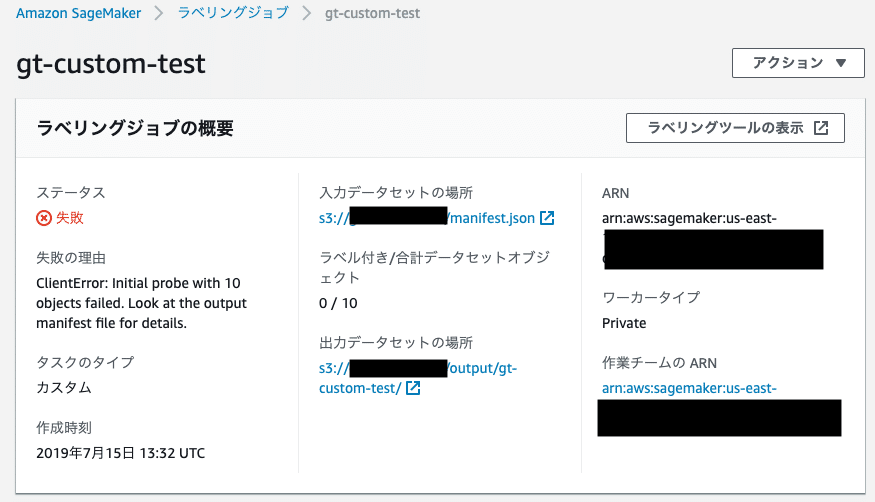
指定したメールアドレスにこんなメールが届きます。
リンク開いてログイン
全てのタスクが終わった扱いになっています。
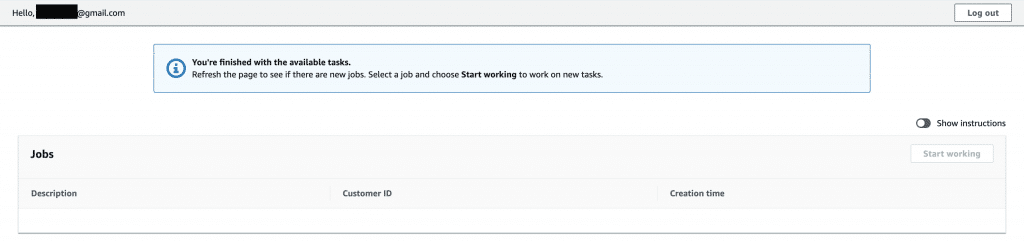
改めてSageMakerに戻って、ラベリングツールの表示をクリック。

やはりどうやらS3のファイルが読み込めていないようです。
さてローカルからS3のファイルにアクセスできたので、ファイル自体がないということはありません。
思いついた原因は2つ。
- SageMakerにS3へのアクセス権がない
- SageMakerにawsブログで指定されたS3へのアクセス権がない
後者が疑わしかったので適当なダミーファイルを自分のS3バケットに作成。マニフェストファイルを書き換えて再度ラベリングジョブを作成。
SageMakerコンソールからラベリングツールの表示を選択。見えた。
右上のテキストの方はなぜか上手く取れてませんがだいたいわかりました。
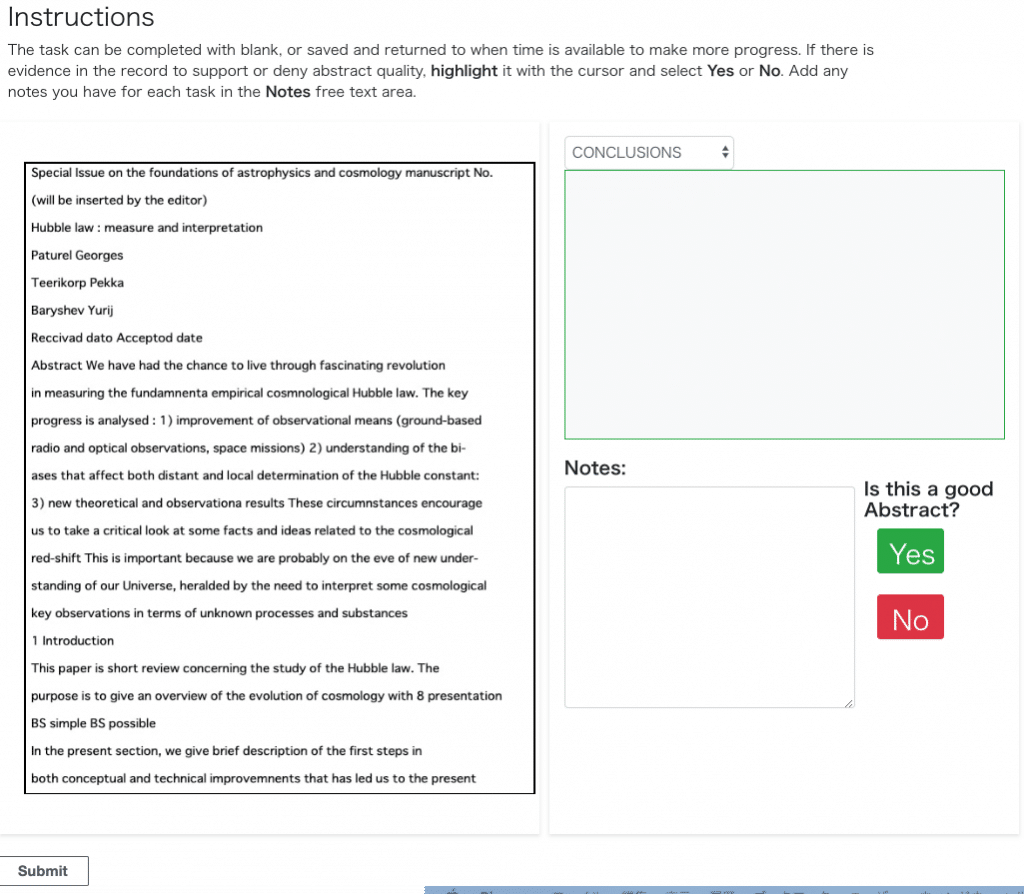
ワーカーとしてラベリングジョブを確認。こっちはテキストを確認できました。
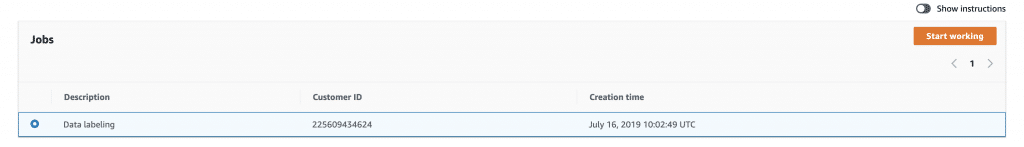

テキストを選択したら色付けされました。

試しにsubmit。どんな出力になるか確認しましょう。
出力先のoutput.manifestを見ると、source, text, metadataは入力に使ったマニフェストファイルと同じ。
その後ワーカーごとにworkerIdとannotationDataが入っています。どうやらトークン単位でアノテーションの範囲を表現しているようです。ここら辺詳しくはserver/processing/sagemaker-gt-postprocess.pyに定義してあります。ここをいじれば任意の形で出力できそうですね。

という感じでなんだかんだ再現するのに丸二日かかってしまいました。実際やってみると上手くいかないことたくさんあるよね。
デフォルトのマニュフェストファイルで実行できた人いたら教えてください。
余談
上手くいくまで3回かかりました。
全てのタスクが完了したらステータスが完了になります。こうなったら停止しなくていいみたい。
何を基準に料金が発生するか、どれくらいになるかは試算しときたいですね。
あと、タスク完了後にS3のマニュフェストファイルを差し替えたけど、タスクは更新されませんでした。大人しくクローンして作り直す必要がありそうです。
これからpre, postのLambdaによる処理、Reactで作られたweb部分を自分用に改造していきます。