ScispaCyでsentence split
論文記事のsentence split問題
一番どシンプルにsentence splitを行うなら、ピリオド(日本語なら句点)で区切ることだ
目次
問題
しかし論文ではこんな感じで、’i.p.’を文末と認識して分割してしまう(i.p.はintraperitoneal = 腹腔投与)

他にも組織名とか

No. でブチギレる

画像は全てtestから
ScispaCy
この悩みを投稿したところTwitterで親切な方に教えていただいた
spaCyは聞いたことあったけど、science特化のものがあったとは。開発元はAI2
やっぱすげえやAI2は!
簡単に使ってみます。
pip install spacy
なんかエラー
import spacy
nlp = spacy.load("en_core_sci_sm")
doc = nlp("Alterations in the hypocretin receptor 2 and preprohypocretin genes produce narcolepsy in some animals.")
OSError: [E050] Can't find model 'en_core_sci_sm'. It doesn't seem to be a shortcut link, a Python package or a valid path to a data directory.
どうやらモデルをダウンロードする必要があるみたい(spaCy初心者並感)
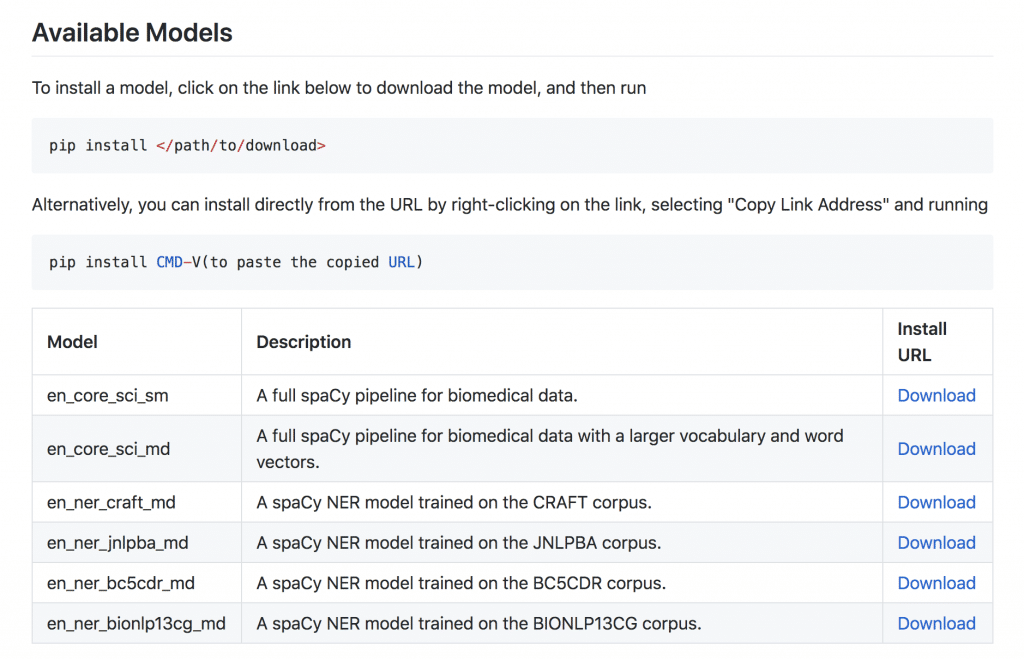
各モデルの性能はこんな感じ
sci_mdの方が若干性能が高いけど、”larger vocabulary and word vectors”を使っているとのことでその分時間かかりそう
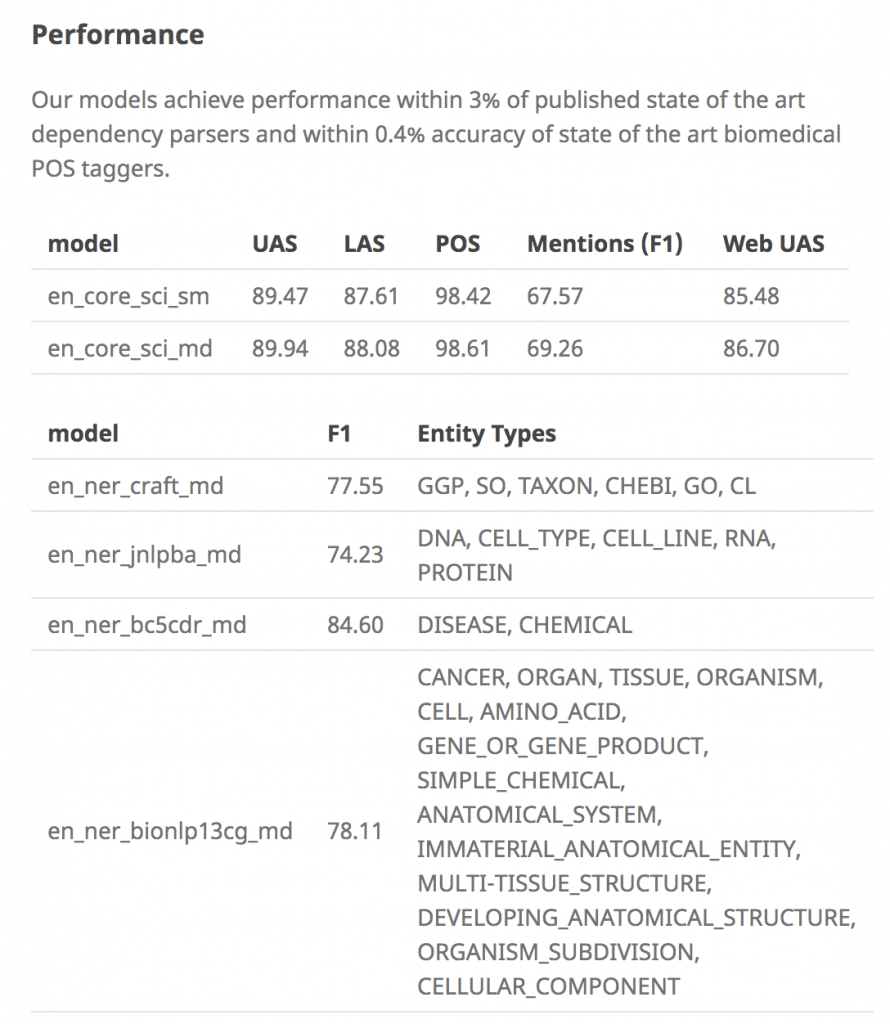
既存タスクとの比較も見たいですね、要文献調査
話は戻って、GithubのDownloadボタンクリックして.tag.gzファイルを取得(圧縮ファイルを指定してpipできるのを初めて知った)
pip install /path/to/xxx.tar.gz
ちなみにどうやらspaCyとscispaCyのバージョンを調整しないとエラーが出るらしい
My guess is that you have an incompatible version for the model that you downloaded. You’ll want to make sure that for
Error when trying to load “en_core_sci_sm”scispacyversion0.1.0you havespacyversion2.0.18and use models that have0.1.0in the url, like this onehttps://s3-us-west-2.amazonaws.com/ai2-s2-scispacy/releases/v0.1.0/en_core_sci_sm-0.1.0.tar.gz. We just upgradedscispacyto version0.2.0which is compatible withspacyversion2.1.3and requires models with0.2.0in the url, like this onehttps://s3-us-west-2.amazonaws.com/ai2-s2-scispacy/releases/v0.2.0/en_core_sci_sm-0.2.0.tar.gz. The README has been updated to have model links forscispacyversion0.2.0as of yesterday.
手元でバージョンチェック
scispacy 0.2.0とspacy 2.1.3がセットらしい
僕の環境に入っているのは0.2.2と2.1.4、大丈夫そう。多分
pip freeze | grep 'spacy'
scispacy==0.2.2
spacy==2.1.4
試しにentityを抜いてみる
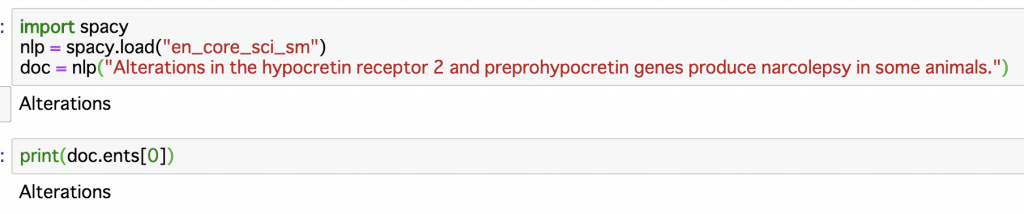
なるほど
基本的にはspacyの機能で、科学/バイオに特化させたモデルを読み込んで使うみたい
つまりは諸々の作業、sentence splitなどはspaCyを参照すれば良さそうです
一方でscispaCy固有の機能もあるみたい
略語の定義を検知したり、他にも開発中っぽい

sentence split
で。求めていたsentence splitはどうなんでしょうか
doc.sents()とやれば文分割してくれる
こんな感じ

で肝心の文章はどうなったかというと、
OK: 投与経路: s41598-017-01804-2

NG: 組織名: s41598-017-06888-4

OK: 番号: s41598-018-25987-4

組織名以外は綺麗になった!伊達にバイオ特化を謳っていませんね!
しかし、独自に追加した表現で引っかかってしまった。nltkでは平気だったんですが
画像はhtmlのiタグの情報をテキストに埋め込むためにつけたprefix, suffixで分割が起きている

もう一点課題は、時間がかかること
1629本の論文に対して、nltkだと2秒ですんだのが、scispaCyだと20分かかった
600倍!
イテレーション回す初期段階で使うより、最後に綺麗なデータを作るときに使おうと思います
いいライブラリでした
情報ありがとうございました!
こんな感じで、バイオ論文の自然言語処理やってます
楽しいですね
興味ある方はぜひ声をかけてください